3.4. 演習:内視鏡画像診断#
内視鏡を利用した胃がん検査は、X 線検査に比べて精度が高く、がんの早期発見に大きく貢献しています。しかし、内視鏡検査では医師が画像を確認しながら診断を行う必要があり、その作業負担が非常に大きいという課題があります。近年、人工知能(AI)を活用した疾患部位の自動検出技術が進展しており、これにより医師の負担軽減や診断の効率化が期待されています。本節では、深層ニューラルネットワークを用いて内視鏡画像から胃腸疾患を診断するプログラムの開発方法を学びます。この演習を通じて、深層学習を活用した診断支援の可能性を具体的に体験し、実際の医療応用を意識した知識とスキルを深めることを目指します。
3.4.1. 演習準備#
3.4.1.1. ライブラリ#
本節で利用するライブラリを読み込みます。NumPy、Pnadas、Matplotlib、Pillow(PIL)などのライブライは、モデルの性能や推論結果などの可視化に利用します。scikit-learn(sklearn)、PyTorch(torch)、torchvision は機械学習関連のライブラリであり、モデルの構築、検証や推論などに利用します。また、OpenCV(cv2)、pytorch_grad_cam(grad-cam)などはモデルの推論根拠を可視化するために利用します。
# visualization
import numpy as np
import pandas as pd
import matplotlib.pyplot as plt
import PIL.Image
# machine learning
import sklearn.metrics
import torch
import torchvision
# grad-CAM visualization
import cv2
import pytorch_grad_cam
print(f'torch v{torch.__version__}; torchvision v{torchvision.__version__}')
torch v2.8.0+cu126; torchvision v0.23.0+cu126
ライブラリの読み込み時に ImportError や ModuleNotFoundError が発生した場合は、該当するライブラリをインストールしてください。ライブラリのバージョンを揃える必要はありませんが、PyTorch(torch)および torchvision が上記のバージョンと異なる時、実行中に警告メッセージが現れたり、同じ結果にならなかったりする可能性があります。
3.4.1.2. データセット#
本節では、Simula Research Laboratory によって公開されている Kvasir データセット[1]を使用します。このデータセットは、内視鏡画像を集めた医療用データセットであり、研究および教育目的に限り利用が許可されています[2]。
Kvasir データセットは 8 つのカテゴリに分類されていますが、本節ではその中の健全な盲腸(normal-cecum)、健全な幽門(normal-pylorus)、健全な食道胃粘膜移行帯(normal-z-line)、食道炎(esophagitis)、潰瘍性大腸炎(ulcerative-colitis)、ポリープ(polyps)の 6 カテゴリを対象に取り扱います(Fig. 3.12)。
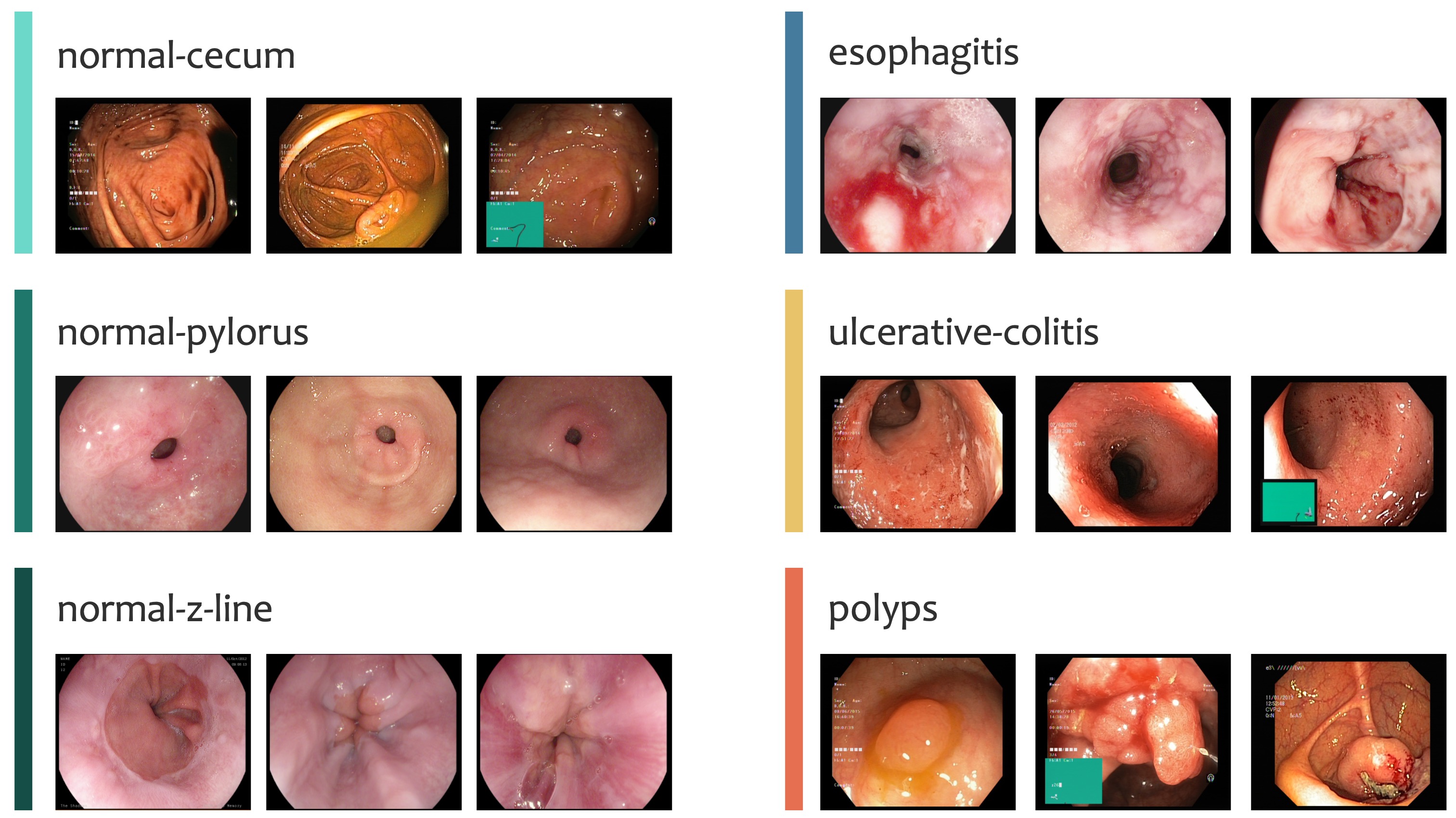
Fig. 3.12 Kvasir データセットに含まれる各カテゴリのサンプル画像。#
本節では、プログラムを短時間で実行できるようにするため、オリジナルの Kvasir データセットから各カテゴリごとにランダムで 100 枚の訓練画像、20 枚の検証画像、20 枚のテスト画像を抽出し、小規模なデータセットを作成して使用します。Jupyter Notebook 上では、以下のコマンドを実行することでデータセットをダウンロードできます。
!wget https://dl.biopapyrus.jp/data/kvasir.zip
!unzip kvasir.zip
3.4.1.3. 画像前処理#
畳み込みニューラルネットワークは、ニューロンの数などが固定されているため、入力する画像のサイズにも制限があります。例えば、本節で使用する DenseNet 121 [3] では、224×224 ピクセルの正方形画像を入力として設計されています。また、PyTorch ではすべてのデータをテンソル形式で扱う必要があります。そのため、畳み込みニューラルネットワークに画像を入力する前に、画像サイズを適切に調整し、テンソル型に変換するといった前処理を行う必要があります。以下では、この前処理の手順を定義します。
class SquareResize:
def __init__(self, shape=224, bg_color = (0, 0, 0)):
self.shape = shape
self.bg_color = tuple(bg_color)
def __call__(self, img):
w, h = img.size
img_square = None
if w == h:
img_square = img
elif w > h:
img_square = PIL.Image.new(img.mode, (w, w), self.bg_color)
img_square.paste(img, (0, (w - h) // 2))
else:
img_square = PIL.Image.new(img.mode, (h, h), self.bg_color)
img_square.paste(img, ((h - w) // 2, 0))
img_square = img_square.resize((self.shape, self.shape))
return img_square
transform = torchvision.transforms.Compose([
SquareResize(),
torchvision.transforms.ToTensor(),
torchvision.transforms.Normalize([0.485, 0.456, 0.406], [0.229, 0.224, 0.225])
])
画像データは通常、0 から 255 の範囲の整数値で表現されていますが、前処理の段階でこれを正規化します。正規化により、画像データの値は平均約 0.5、標準偏差約 0.23 の範囲に変換され、モデルの学習を効率的に進めることができます。なお、正規化の際に平均を 0.50、分散を 0.23 のような切りの良い数値にしない理由は、これから利用する torchvision.models が提供する訓練済みモデルが、特定の数値(例えば平均 0.485、標準偏差 0.229)で訓練されているためです。そのため、この訓練済みモデルに合わせて正規化を行います。
3.4.1.4. 計算デバイス#
計算を行うデバイスを設定します。PyTorch が GPU を認識できる場合は GPU を利用し、認識できない場合は CPU を使用するように設定します。
device = torch.device('cuda') if torch.cuda.is_available() else torch.device('cpu')
3.4.2. モデル構築#
本節では、物体分類のアーキテクチャとして DenseNet 121 を使用します。このアーキテクチャは、深い層を持ちながらもパラメータ数を大幅に削減した設計が特徴です。
torchvision.models モジュールで提供されている DenseNet 121 は、飛行機や車、人など、1000 種類の一般的な物体を分類するように設計されています。これに対して、本節では、normal-cecum、normal-pylorus、normal-z-line、esophagitis、ulcerative-colitis、polyps の 6 カテゴリの分類問題を扱います。そのため、torchvision.models モジュールから読み込んだ DenseNet 121 の出力層のユニット数を 6 に変更する必要があります。この修正作業はアーキテクチャを呼び出すたびに行う必要があり、手間がかかります。そこで、一連の処理を関数化してから利用します。
def densenet121(num_classes, weights=None):
model = torchvision.models.densenet121(weights='DEFAULT')
in_features = model.classifier.in_features
model.classifier = torch.nn.Linear(in_features, num_classes)
if weights is not None:
model.load_state_dict(torch.load(weights))
return model
model = densenet121(6)
3.4.3. モデル訓練#
モデルが学習データを効率よく学習できるようにするため、損失関数(criterion)、学習アルゴリズム(optimizer)、学習率(lr)、および学習率を調整するスケジューラ(lr_scheduler)を設定します。
criterion = torch.nn.CrossEntropyLoss()
optimizer = torch.optim.Adam(model.parameters(), lr=0.0001)
lr_scheduler = torch.optim.lr_scheduler.StepLR(optimizer, step_size=3, gamma=0.1)
次に、訓練データと検証データを読み込み、モデルが入力できる形式に整えます。
train_dataset = torchvision.datasets.ImageFolder('kvasir/train', transform=transform)
train_loader = torch.utils.data.DataLoader(train_dataset, batch_size=32, shuffle=True)
valid_dataset = torchvision.datasets.ImageFolder('kvasir/valid', transform=transform)
valid_loader = torch.utils.data.DataLoader(valid_dataset, batch_size=32, shuffle=False)
準備が整ったら、訓練を開始します。訓練プロセスでは、訓練と検証を交互に繰り返します。訓練では、訓練データを使ってモデルのパラメータを更新し、その際の損失(誤差)を記録します。検証では、検証データを使ってモデルの予測性能(正解率)を計算し、その結果を記録します。このサイクルを繰り返すことで、モデルの精度を少しずつ向上させていきます。
model.to(device)
num_epochs = 10
metric_dict = []
for epoch in range(num_epochs):
# training phase
model.train()
running_loss = 0.0
n_correct_train = 0
n_train_samples = 0
for images, labels in train_loader:
images = images.to(device)
labels = labels.to(device)
outputs = model(images)
loss = criterion(outputs, labels)
_, predicted_labels = torch.max(outputs.data, 1)
n_correct_train += torch.sum(predicted_labels == labels).item()
n_train_samples += labels.size(0)
running_loss += loss.item() / len(train_loader)
optimizer.zero_grad()
loss.backward()
optimizer.step()
lr_scheduler.step()
# validation phase
model.eval()
n_correct_valid = 0
n_valid_samples = 0
with torch.no_grad():
for images, labels in valid_loader:
images = images.to(device)
labels = labels.to(device)
outputs = model(images)
_, predicted_labels = torch.max(outputs.data, 1)
n_correct_valid += torch.sum(predicted_labels == labels).item()
n_valid_samples += labels.size(0)
metric_dict.append({
'epoch': epoch + 1,
'train_loss': running_loss,
'train_acc': n_correct_train / n_train_samples,
'valid_acc': n_correct_valid / n_valid_samples
})
print(metric_dict[-1])
Show code cell output
{'epoch': 1, 'train_loss': 0.9828014860027714, 'train_acc': 0.7083333333333334, 'valid_acc': 0.9}
{'epoch': 2, 'train_loss': 0.2488036853702445, 'train_acc': 0.9583333333333334, 'valid_acc': 0.925}
{'epoch': 3, 'train_loss': 0.11977902976305861, 'train_acc': 0.9766666666666667, 'valid_acc': 0.9083333333333333}
{'epoch': 4, 'train_loss': 0.0683305747807026, 'train_acc': 0.995, 'valid_acc': 0.9166666666666666}
{'epoch': 5, 'train_loss': 0.05241301734196513, 'train_acc': 0.9983333333333333, 'valid_acc': 0.9083333333333333}
{'epoch': 6, 'train_loss': 0.06790350828515856, 'train_acc': 1.0, 'valid_acc': 0.9166666666666666}
{'epoch': 7, 'train_loss': 0.05297867845939962, 'train_acc': 1.0, 'valid_acc': 0.9166666666666666}
{'epoch': 8, 'train_loss': 0.045864293057667586, 'train_acc': 0.9983333333333333, 'valid_acc': 0.9166666666666666}
{'epoch': 9, 'train_loss': 0.04347682567803483, 'train_acc': 1.0, 'valid_acc': 0.9166666666666666}
{'epoch': 10, 'train_loss': 0.0528255916739765, 'train_acc': 1.0, 'valid_acc': 0.9083333333333333}
訓練データに対する損失と検証データに対する正解率を可視化し、訓練過程を評価します。
Show code cell source
metric_dict = pd.DataFrame(metric_dict)
fig, ax = plt.subplots(1, 2)
ax[0].plot(metric_dict['epoch'], metric_dict['train_loss'])
ax[0].set_xlabel('epoch')
ax[0].set_ylabel('loss')
ax[0].set_title('Train')
ax[1].plot(metric_dict['epoch'], metric_dict['valid_acc'])
ax[1].set_ylim(0, 1)
ax[1].set_xlabel('epoch')
ax[1].set_ylabel('accuracy')
ax[1].set_title('Validation')
plt.tight_layout()
fig.show()
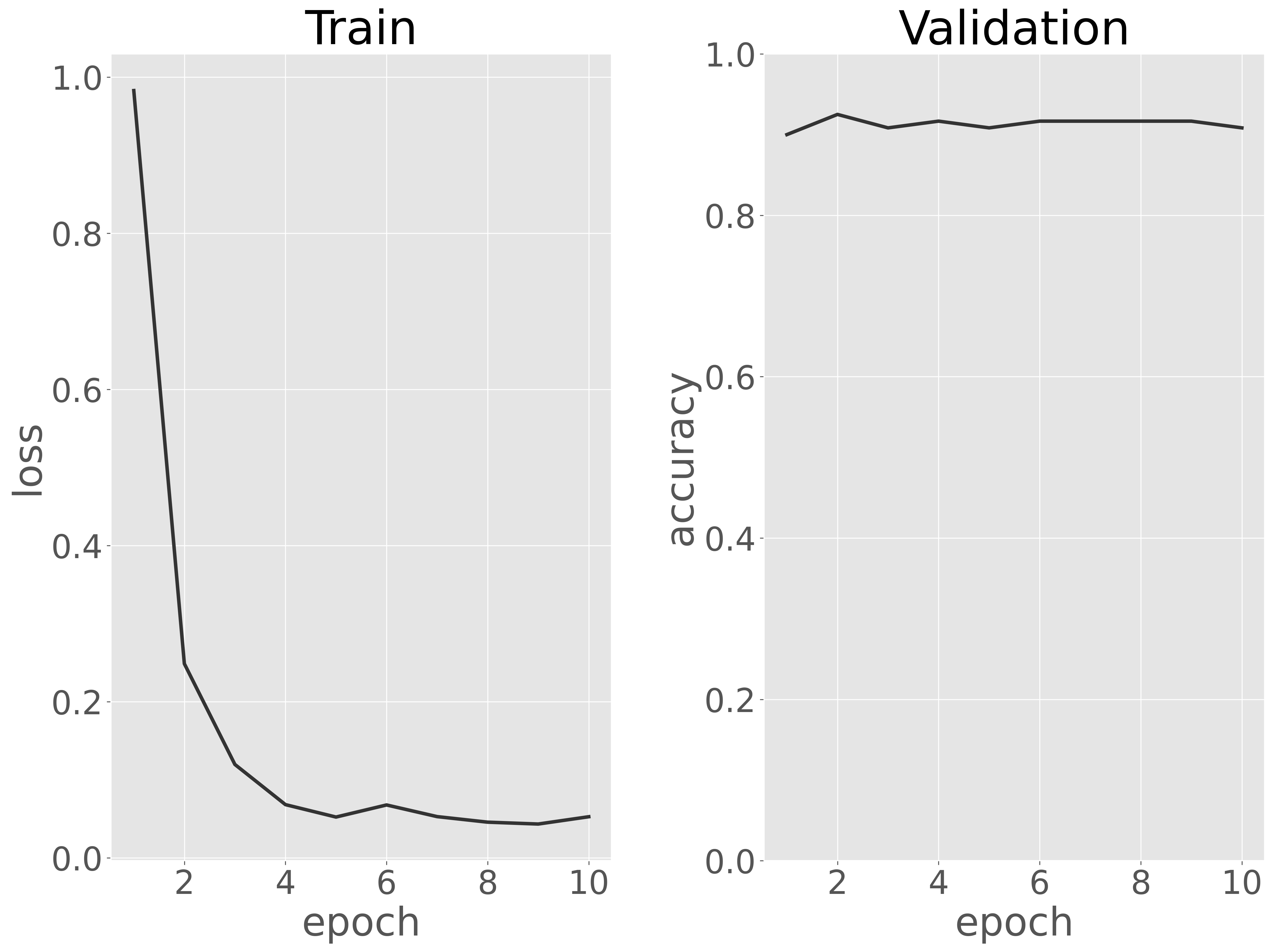
可視化の結果から、エポックが進むにつれて訓練データに対する損失は減少し、7 エポック以降に収束し始める傾向が見られました。また、検証データに対する正解率は、最初の数エポックで約 に達した後、エポック数が増えてもそれ以上の向上は見られませんでした。このグラフから、数エポックの訓練だけで最適なモデルが得られることがわかります。
次に、同じ手順を他の深層ニューラルネットワークアーキテクチャ(ResNet や Inception など)に対して実施し、それぞれの検証性能を比較します。そして、このデータセットに最適なアーキテクチャを選択します。ただし、本節ではモデル(アーキテクチャ)選択を行わずに、DenseNet 121 を最適なアーキテクチャとして採用し、次のステップに進みます。
次のステップでは、訓練サブセットと検証サブセットを統合し、最適なアーキテクチャで最初から訓練を行います。
!mkdir kvasir/trainvalid
!cp -r kvasir/train/* kvasir/trainvalid
!cp -r kvasir/valid/* kvasir/trainvalid
最適なモデルを選択する段階で、数エポックの訓練だけでも十分に高い予測性能を獲得できたことがわかったので、ここでは訓練サブセットと検証サブセットを統合したデータに対して 5 エポックだけ訓練させます。
# model
model = densenet121(6)
model.to(device)
# training params
criterion = torch.nn.CrossEntropyLoss()
optimizer = torch.optim.Adam(model.parameters(), lr=0.0001)
lr_scheduler = torch.optim.lr_scheduler.StepLR(optimizer, step_size=3, gamma=0.1)
# training data
train_dataset = torchvision.datasets.ImageFolder('kvasir/trainvalid', transform=transform)
train_loader = torch.utils.data.DataLoader(train_dataset, batch_size=32, shuffle=True)
# training
num_epochs = 5
metric_dict = []
for epoch in range(num_epochs):
model.train()
running_loss = 0.0
n_correct_train = 0
n_train_samples = 0
for images, labels in train_loader:
images = images.to(device)
labels = labels.to(device)
outputs = model(images)
loss = criterion(outputs, labels)
_, predicted_labels = torch.max(outputs.data, 1)
n_correct_train += torch.sum(predicted_labels == labels).item()
n_train_samples += labels.size(0)
running_loss += loss.item() / len(train_loader)
optimizer.zero_grad()
loss.backward()
optimizer.step()
lr_scheduler.step()
metric_dict.append({
'epoch': epoch + 1,
'train_loss': running_loss,
'train_acc': n_correct_train / n_train_samples,
})
print(metric_dict[-1])
Show code cell output
{'epoch': 1, 'train_loss': 0.9458750991717628, 'train_acc': 0.7041666666666667}
{'epoch': 2, 'train_loss': 0.2610346535625665, 'train_acc': 0.9458333333333333}
{'epoch': 3, 'train_loss': 0.10303420443897661, 'train_acc': 0.9861111111111112}
{'epoch': 4, 'train_loss': 0.060086415513702064, 'train_acc': 0.9958333333333333}
{'epoch': 5, 'train_loss': 0.05524141989324405, 'train_acc': 0.9986111111111111}
訓練が完了したら、訓練済みモデルの重みをファイルに保存します。
model = model.to('cpu')
torch.save(model.state_dict(), 'kvasir.pth')
3.4.4. モデル評価#
最適なモデルが得られたら、次にテストデータを用いてモデルを詳細に評価します。正解率だけでなく、適合率、再現率、F1 スコアなどの評価指標を計算し、モデルを総合的に評価します。まず、テストデータをモデルに入力し、その予測結果を取得します。
model = densenet121(6, 'kvasir.pth')
model.to(device)
model.eval()
test_dataset = torchvision.datasets.ImageFolder('kvasir/test', transform=transform)
test_loader = torch.utils.data.DataLoader(test_dataset, batch_size=32, shuffle=False)
pred_labels = []
true_labels = []
with torch.no_grad():
for images, labels in test_loader:
images = images.to(device)
labels = labels.to(device)
outputs = model(images)
_, _labels = torch.max(outputs.data, 1)
#print(_labels)
pred_labels.extend(_labels.cpu().detach().numpy().tolist())
true_labels.extend(labels.cpu().detach().numpy().tolist())
pred_labels = [test_dataset.classes[_] for _ in pred_labels]
true_labels = [test_dataset.classes[_] for _ in true_labels]
次に、予測結果とラベルを比較し、混同行列を作成します。これにより、間違いやすいカテゴリを特定することができます。
cm = sklearn.metrics.confusion_matrix(true_labels, pred_labels)
cm
array([[13, 0, 0, 7, 0, 0],
[ 0, 20, 0, 0, 0, 0],
[ 0, 0, 20, 0, 0, 0],
[ 2, 0, 0, 18, 0, 0],
[ 0, 2, 0, 0, 18, 0],
[ 1, 0, 0, 0, 1, 18]])
cmp = sklearn.metrics.ConfusionMatrixDisplay(cm, display_labels=test_dataset.classes)
cmp.plot(xticks_rotation='vertical')
<sklearn.metrics._plot.confusion_matrix.ConfusionMatrixDisplay at 0x7f619082ddd0>
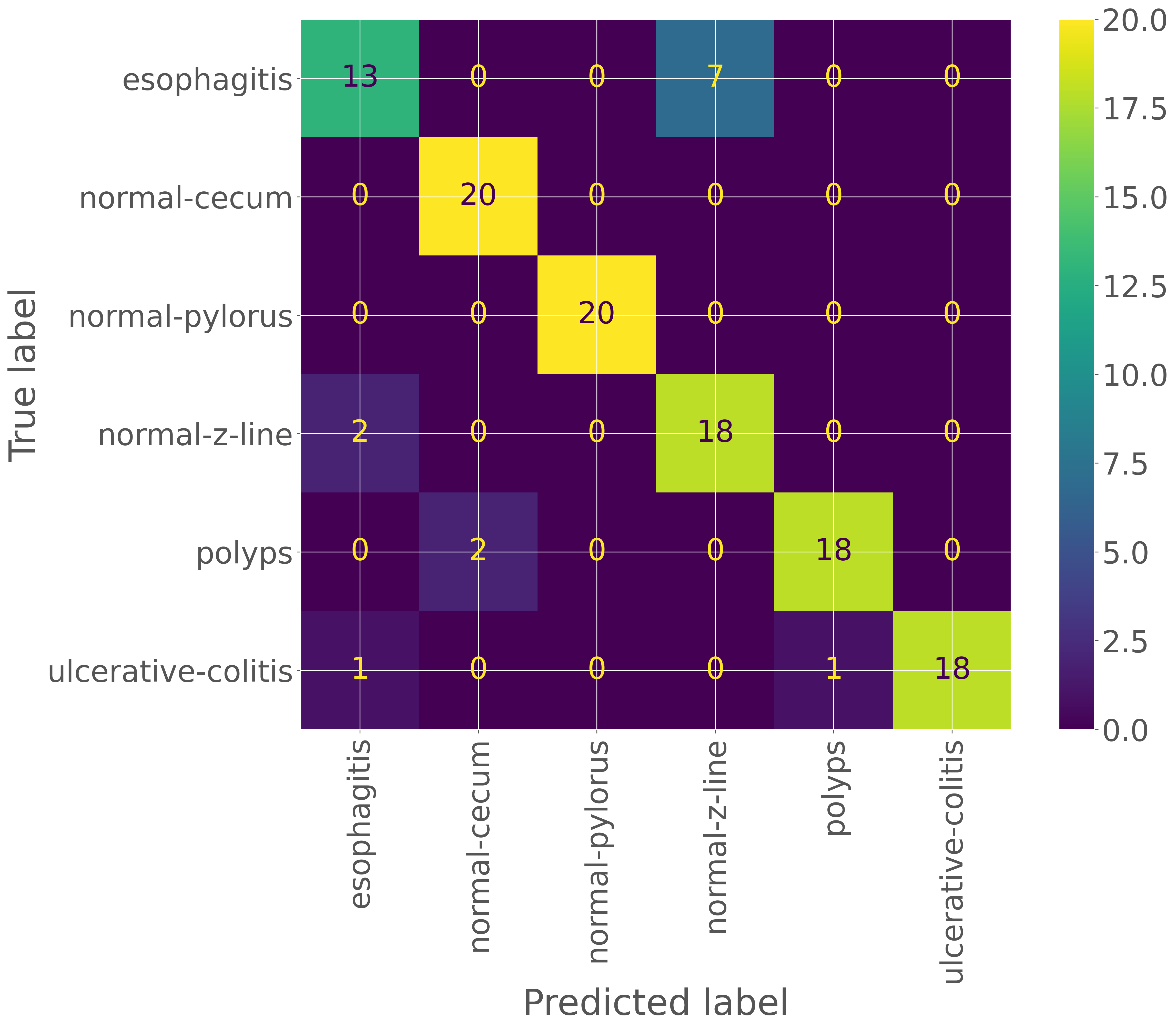
それぞれのクラスに対する適合率、再現率、F1 スコアなどは、scikit-learn ライブラリを利用して計算します。
pd.DataFrame(sklearn.metrics.classification_report(true_labels, pred_labels, output_dict=True))
| esophagitis | normal-cecum | normal-pylorus | normal-z-line | polyps | ulcerative-colitis | accuracy | macro avg | weighted avg | |
|---|---|---|---|---|---|---|---|---|---|
| precision | 0.812500 | 0.909091 | 1.0 | 0.72 | 0.947368 | 1.000000 | 0.891667 | 0.898160 | 0.898160 |
| recall | 0.650000 | 1.000000 | 1.0 | 0.90 | 0.900000 | 0.900000 | 0.891667 | 0.891667 | 0.891667 |
| f1-score | 0.722222 | 0.952381 | 1.0 | 0.80 | 0.923077 | 0.947368 | 0.891667 | 0.890841 | 0.890841 |
| support | 20.000000 | 20.000000 | 20.0 | 20.00 | 20.000000 | 20.000000 | 0.891667 | 120.000000 | 120.000000 |
3.4.5. 推論#
推論を行う際には、訓練や評価時と同様に、torchvision.models モジュールから DenseNet 121 のアーキテクチャを読み込み、出力層のクラス数を設定します。その後、load_state_dict メソッドを使用して訓練済みの重みファイルをモデルにロードします。これらの処理はすでに関数化(densenet121)されているため、その関数を利用して簡単に実行できます。
labels = ['esophagitis', 'normal-cecum', 'normal-pylorus', 'normal-z-line', 'polyps', 'ulcerative-colitis']
model = densenet121(6, 'kvasir.pth')
model.to(device)
model.eval()
Show code cell output
DenseNet(
(features): Sequential(
(conv0): Conv2d(3, 64, kernel_size=(7, 7), stride=(2, 2), padding=(3, 3), bias=False)
(norm0): BatchNorm2d(64, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu0): ReLU(inplace=True)
(pool0): MaxPool2d(kernel_size=3, stride=2, padding=1, dilation=1, ceil_mode=False)
(denseblock1): _DenseBlock(
(denselayer1): _DenseLayer(
(norm1): BatchNorm2d(64, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu1): ReLU(inplace=True)
(conv1): Conv2d(64, 128, kernel_size=(1, 1), stride=(1, 1), bias=False)
(norm2): BatchNorm2d(128, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu2): ReLU(inplace=True)
(conv2): Conv2d(128, 32, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1), bias=False)
)
(denselayer2): _DenseLayer(
(norm1): BatchNorm2d(96, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu1): ReLU(inplace=True)
(conv1): Conv2d(96, 128, kernel_size=(1, 1), stride=(1, 1), bias=False)
(norm2): BatchNorm2d(128, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu2): ReLU(inplace=True)
(conv2): Conv2d(128, 32, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1), bias=False)
)
(denselayer3): _DenseLayer(
(norm1): BatchNorm2d(128, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu1): ReLU(inplace=True)
(conv1): Conv2d(128, 128, kernel_size=(1, 1), stride=(1, 1), bias=False)
(norm2): BatchNorm2d(128, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu2): ReLU(inplace=True)
(conv2): Conv2d(128, 32, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1), bias=False)
)
(denselayer4): _DenseLayer(
(norm1): BatchNorm2d(160, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu1): ReLU(inplace=True)
(conv1): Conv2d(160, 128, kernel_size=(1, 1), stride=(1, 1), bias=False)
(norm2): BatchNorm2d(128, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu2): ReLU(inplace=True)
(conv2): Conv2d(128, 32, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1), bias=False)
)
(denselayer5): _DenseLayer(
(norm1): BatchNorm2d(192, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu1): ReLU(inplace=True)
(conv1): Conv2d(192, 128, kernel_size=(1, 1), stride=(1, 1), bias=False)
(norm2): BatchNorm2d(128, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu2): ReLU(inplace=True)
(conv2): Conv2d(128, 32, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1), bias=False)
)
(denselayer6): _DenseLayer(
(norm1): BatchNorm2d(224, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu1): ReLU(inplace=True)
(conv1): Conv2d(224, 128, kernel_size=(1, 1), stride=(1, 1), bias=False)
(norm2): BatchNorm2d(128, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu2): ReLU(inplace=True)
(conv2): Conv2d(128, 32, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1), bias=False)
)
)
(transition1): _Transition(
(norm): BatchNorm2d(256, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu): ReLU(inplace=True)
(conv): Conv2d(256, 128, kernel_size=(1, 1), stride=(1, 1), bias=False)
(pool): AvgPool2d(kernel_size=2, stride=2, padding=0)
)
(denseblock2): _DenseBlock(
(denselayer1): _DenseLayer(
(norm1): BatchNorm2d(128, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu1): ReLU(inplace=True)
(conv1): Conv2d(128, 128, kernel_size=(1, 1), stride=(1, 1), bias=False)
(norm2): BatchNorm2d(128, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu2): ReLU(inplace=True)
(conv2): Conv2d(128, 32, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1), bias=False)
)
(denselayer2): _DenseLayer(
(norm1): BatchNorm2d(160, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu1): ReLU(inplace=True)
(conv1): Conv2d(160, 128, kernel_size=(1, 1), stride=(1, 1), bias=False)
(norm2): BatchNorm2d(128, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu2): ReLU(inplace=True)
(conv2): Conv2d(128, 32, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1), bias=False)
)
(denselayer3): _DenseLayer(
(norm1): BatchNorm2d(192, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu1): ReLU(inplace=True)
(conv1): Conv2d(192, 128, kernel_size=(1, 1), stride=(1, 1), bias=False)
(norm2): BatchNorm2d(128, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu2): ReLU(inplace=True)
(conv2): Conv2d(128, 32, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1), bias=False)
)
(denselayer4): _DenseLayer(
(norm1): BatchNorm2d(224, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu1): ReLU(inplace=True)
(conv1): Conv2d(224, 128, kernel_size=(1, 1), stride=(1, 1), bias=False)
(norm2): BatchNorm2d(128, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu2): ReLU(inplace=True)
(conv2): Conv2d(128, 32, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1), bias=False)
)
(denselayer5): _DenseLayer(
(norm1): BatchNorm2d(256, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu1): ReLU(inplace=True)
(conv1): Conv2d(256, 128, kernel_size=(1, 1), stride=(1, 1), bias=False)
(norm2): BatchNorm2d(128, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu2): ReLU(inplace=True)
(conv2): Conv2d(128, 32, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1), bias=False)
)
(denselayer6): _DenseLayer(
(norm1): BatchNorm2d(288, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu1): ReLU(inplace=True)
(conv1): Conv2d(288, 128, kernel_size=(1, 1), stride=(1, 1), bias=False)
(norm2): BatchNorm2d(128, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu2): ReLU(inplace=True)
(conv2): Conv2d(128, 32, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1), bias=False)
)
(denselayer7): _DenseLayer(
(norm1): BatchNorm2d(320, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu1): ReLU(inplace=True)
(conv1): Conv2d(320, 128, kernel_size=(1, 1), stride=(1, 1), bias=False)
(norm2): BatchNorm2d(128, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu2): ReLU(inplace=True)
(conv2): Conv2d(128, 32, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1), bias=False)
)
(denselayer8): _DenseLayer(
(norm1): BatchNorm2d(352, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu1): ReLU(inplace=True)
(conv1): Conv2d(352, 128, kernel_size=(1, 1), stride=(1, 1), bias=False)
(norm2): BatchNorm2d(128, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu2): ReLU(inplace=True)
(conv2): Conv2d(128, 32, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1), bias=False)
)
(denselayer9): _DenseLayer(
(norm1): BatchNorm2d(384, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu1): ReLU(inplace=True)
(conv1): Conv2d(384, 128, kernel_size=(1, 1), stride=(1, 1), bias=False)
(norm2): BatchNorm2d(128, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu2): ReLU(inplace=True)
(conv2): Conv2d(128, 32, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1), bias=False)
)
(denselayer10): _DenseLayer(
(norm1): BatchNorm2d(416, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu1): ReLU(inplace=True)
(conv1): Conv2d(416, 128, kernel_size=(1, 1), stride=(1, 1), bias=False)
(norm2): BatchNorm2d(128, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu2): ReLU(inplace=True)
(conv2): Conv2d(128, 32, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1), bias=False)
)
(denselayer11): _DenseLayer(
(norm1): BatchNorm2d(448, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu1): ReLU(inplace=True)
(conv1): Conv2d(448, 128, kernel_size=(1, 1), stride=(1, 1), bias=False)
(norm2): BatchNorm2d(128, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu2): ReLU(inplace=True)
(conv2): Conv2d(128, 32, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1), bias=False)
)
(denselayer12): _DenseLayer(
(norm1): BatchNorm2d(480, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu1): ReLU(inplace=True)
(conv1): Conv2d(480, 128, kernel_size=(1, 1), stride=(1, 1), bias=False)
(norm2): BatchNorm2d(128, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu2): ReLU(inplace=True)
(conv2): Conv2d(128, 32, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1), bias=False)
)
)
(transition2): _Transition(
(norm): BatchNorm2d(512, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu): ReLU(inplace=True)
(conv): Conv2d(512, 256, kernel_size=(1, 1), stride=(1, 1), bias=False)
(pool): AvgPool2d(kernel_size=2, stride=2, padding=0)
)
(denseblock3): _DenseBlock(
(denselayer1): _DenseLayer(
(norm1): BatchNorm2d(256, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu1): ReLU(inplace=True)
(conv1): Conv2d(256, 128, kernel_size=(1, 1), stride=(1, 1), bias=False)
(norm2): BatchNorm2d(128, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu2): ReLU(inplace=True)
(conv2): Conv2d(128, 32, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1), bias=False)
)
(denselayer2): _DenseLayer(
(norm1): BatchNorm2d(288, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu1): ReLU(inplace=True)
(conv1): Conv2d(288, 128, kernel_size=(1, 1), stride=(1, 1), bias=False)
(norm2): BatchNorm2d(128, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu2): ReLU(inplace=True)
(conv2): Conv2d(128, 32, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1), bias=False)
)
(denselayer3): _DenseLayer(
(norm1): BatchNorm2d(320, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu1): ReLU(inplace=True)
(conv1): Conv2d(320, 128, kernel_size=(1, 1), stride=(1, 1), bias=False)
(norm2): BatchNorm2d(128, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu2): ReLU(inplace=True)
(conv2): Conv2d(128, 32, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1), bias=False)
)
(denselayer4): _DenseLayer(
(norm1): BatchNorm2d(352, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu1): ReLU(inplace=True)
(conv1): Conv2d(352, 128, kernel_size=(1, 1), stride=(1, 1), bias=False)
(norm2): BatchNorm2d(128, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu2): ReLU(inplace=True)
(conv2): Conv2d(128, 32, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1), bias=False)
)
(denselayer5): _DenseLayer(
(norm1): BatchNorm2d(384, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu1): ReLU(inplace=True)
(conv1): Conv2d(384, 128, kernel_size=(1, 1), stride=(1, 1), bias=False)
(norm2): BatchNorm2d(128, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu2): ReLU(inplace=True)
(conv2): Conv2d(128, 32, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1), bias=False)
)
(denselayer6): _DenseLayer(
(norm1): BatchNorm2d(416, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu1): ReLU(inplace=True)
(conv1): Conv2d(416, 128, kernel_size=(1, 1), stride=(1, 1), bias=False)
(norm2): BatchNorm2d(128, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu2): ReLU(inplace=True)
(conv2): Conv2d(128, 32, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1), bias=False)
)
(denselayer7): _DenseLayer(
(norm1): BatchNorm2d(448, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu1): ReLU(inplace=True)
(conv1): Conv2d(448, 128, kernel_size=(1, 1), stride=(1, 1), bias=False)
(norm2): BatchNorm2d(128, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu2): ReLU(inplace=True)
(conv2): Conv2d(128, 32, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1), bias=False)
)
(denselayer8): _DenseLayer(
(norm1): BatchNorm2d(480, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu1): ReLU(inplace=True)
(conv1): Conv2d(480, 128, kernel_size=(1, 1), stride=(1, 1), bias=False)
(norm2): BatchNorm2d(128, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu2): ReLU(inplace=True)
(conv2): Conv2d(128, 32, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1), bias=False)
)
(denselayer9): _DenseLayer(
(norm1): BatchNorm2d(512, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu1): ReLU(inplace=True)
(conv1): Conv2d(512, 128, kernel_size=(1, 1), stride=(1, 1), bias=False)
(norm2): BatchNorm2d(128, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu2): ReLU(inplace=True)
(conv2): Conv2d(128, 32, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1), bias=False)
)
(denselayer10): _DenseLayer(
(norm1): BatchNorm2d(544, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu1): ReLU(inplace=True)
(conv1): Conv2d(544, 128, kernel_size=(1, 1), stride=(1, 1), bias=False)
(norm2): BatchNorm2d(128, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu2): ReLU(inplace=True)
(conv2): Conv2d(128, 32, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1), bias=False)
)
(denselayer11): _DenseLayer(
(norm1): BatchNorm2d(576, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu1): ReLU(inplace=True)
(conv1): Conv2d(576, 128, kernel_size=(1, 1), stride=(1, 1), bias=False)
(norm2): BatchNorm2d(128, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu2): ReLU(inplace=True)
(conv2): Conv2d(128, 32, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1), bias=False)
)
(denselayer12): _DenseLayer(
(norm1): BatchNorm2d(608, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu1): ReLU(inplace=True)
(conv1): Conv2d(608, 128, kernel_size=(1, 1), stride=(1, 1), bias=False)
(norm2): BatchNorm2d(128, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu2): ReLU(inplace=True)
(conv2): Conv2d(128, 32, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1), bias=False)
)
(denselayer13): _DenseLayer(
(norm1): BatchNorm2d(640, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu1): ReLU(inplace=True)
(conv1): Conv2d(640, 128, kernel_size=(1, 1), stride=(1, 1), bias=False)
(norm2): BatchNorm2d(128, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu2): ReLU(inplace=True)
(conv2): Conv2d(128, 32, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1), bias=False)
)
(denselayer14): _DenseLayer(
(norm1): BatchNorm2d(672, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu1): ReLU(inplace=True)
(conv1): Conv2d(672, 128, kernel_size=(1, 1), stride=(1, 1), bias=False)
(norm2): BatchNorm2d(128, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu2): ReLU(inplace=True)
(conv2): Conv2d(128, 32, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1), bias=False)
)
(denselayer15): _DenseLayer(
(norm1): BatchNorm2d(704, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu1): ReLU(inplace=True)
(conv1): Conv2d(704, 128, kernel_size=(1, 1), stride=(1, 1), bias=False)
(norm2): BatchNorm2d(128, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu2): ReLU(inplace=True)
(conv2): Conv2d(128, 32, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1), bias=False)
)
(denselayer16): _DenseLayer(
(norm1): BatchNorm2d(736, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu1): ReLU(inplace=True)
(conv1): Conv2d(736, 128, kernel_size=(1, 1), stride=(1, 1), bias=False)
(norm2): BatchNorm2d(128, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu2): ReLU(inplace=True)
(conv2): Conv2d(128, 32, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1), bias=False)
)
(denselayer17): _DenseLayer(
(norm1): BatchNorm2d(768, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu1): ReLU(inplace=True)
(conv1): Conv2d(768, 128, kernel_size=(1, 1), stride=(1, 1), bias=False)
(norm2): BatchNorm2d(128, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu2): ReLU(inplace=True)
(conv2): Conv2d(128, 32, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1), bias=False)
)
(denselayer18): _DenseLayer(
(norm1): BatchNorm2d(800, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu1): ReLU(inplace=True)
(conv1): Conv2d(800, 128, kernel_size=(1, 1), stride=(1, 1), bias=False)
(norm2): BatchNorm2d(128, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu2): ReLU(inplace=True)
(conv2): Conv2d(128, 32, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1), bias=False)
)
(denselayer19): _DenseLayer(
(norm1): BatchNorm2d(832, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu1): ReLU(inplace=True)
(conv1): Conv2d(832, 128, kernel_size=(1, 1), stride=(1, 1), bias=False)
(norm2): BatchNorm2d(128, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu2): ReLU(inplace=True)
(conv2): Conv2d(128, 32, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1), bias=False)
)
(denselayer20): _DenseLayer(
(norm1): BatchNorm2d(864, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu1): ReLU(inplace=True)
(conv1): Conv2d(864, 128, kernel_size=(1, 1), stride=(1, 1), bias=False)
(norm2): BatchNorm2d(128, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu2): ReLU(inplace=True)
(conv2): Conv2d(128, 32, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1), bias=False)
)
(denselayer21): _DenseLayer(
(norm1): BatchNorm2d(896, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu1): ReLU(inplace=True)
(conv1): Conv2d(896, 128, kernel_size=(1, 1), stride=(1, 1), bias=False)
(norm2): BatchNorm2d(128, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu2): ReLU(inplace=True)
(conv2): Conv2d(128, 32, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1), bias=False)
)
(denselayer22): _DenseLayer(
(norm1): BatchNorm2d(928, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu1): ReLU(inplace=True)
(conv1): Conv2d(928, 128, kernel_size=(1, 1), stride=(1, 1), bias=False)
(norm2): BatchNorm2d(128, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu2): ReLU(inplace=True)
(conv2): Conv2d(128, 32, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1), bias=False)
)
(denselayer23): _DenseLayer(
(norm1): BatchNorm2d(960, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu1): ReLU(inplace=True)
(conv1): Conv2d(960, 128, kernel_size=(1, 1), stride=(1, 1), bias=False)
(norm2): BatchNorm2d(128, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu2): ReLU(inplace=True)
(conv2): Conv2d(128, 32, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1), bias=False)
)
(denselayer24): _DenseLayer(
(norm1): BatchNorm2d(992, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu1): ReLU(inplace=True)
(conv1): Conv2d(992, 128, kernel_size=(1, 1), stride=(1, 1), bias=False)
(norm2): BatchNorm2d(128, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu2): ReLU(inplace=True)
(conv2): Conv2d(128, 32, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1), bias=False)
)
)
(transition3): _Transition(
(norm): BatchNorm2d(1024, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu): ReLU(inplace=True)
(conv): Conv2d(1024, 512, kernel_size=(1, 1), stride=(1, 1), bias=False)
(pool): AvgPool2d(kernel_size=2, stride=2, padding=0)
)
(denseblock4): _DenseBlock(
(denselayer1): _DenseLayer(
(norm1): BatchNorm2d(512, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu1): ReLU(inplace=True)
(conv1): Conv2d(512, 128, kernel_size=(1, 1), stride=(1, 1), bias=False)
(norm2): BatchNorm2d(128, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu2): ReLU(inplace=True)
(conv2): Conv2d(128, 32, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1), bias=False)
)
(denselayer2): _DenseLayer(
(norm1): BatchNorm2d(544, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu1): ReLU(inplace=True)
(conv1): Conv2d(544, 128, kernel_size=(1, 1), stride=(1, 1), bias=False)
(norm2): BatchNorm2d(128, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu2): ReLU(inplace=True)
(conv2): Conv2d(128, 32, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1), bias=False)
)
(denselayer3): _DenseLayer(
(norm1): BatchNorm2d(576, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu1): ReLU(inplace=True)
(conv1): Conv2d(576, 128, kernel_size=(1, 1), stride=(1, 1), bias=False)
(norm2): BatchNorm2d(128, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu2): ReLU(inplace=True)
(conv2): Conv2d(128, 32, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1), bias=False)
)
(denselayer4): _DenseLayer(
(norm1): BatchNorm2d(608, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu1): ReLU(inplace=True)
(conv1): Conv2d(608, 128, kernel_size=(1, 1), stride=(1, 1), bias=False)
(norm2): BatchNorm2d(128, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu2): ReLU(inplace=True)
(conv2): Conv2d(128, 32, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1), bias=False)
)
(denselayer5): _DenseLayer(
(norm1): BatchNorm2d(640, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu1): ReLU(inplace=True)
(conv1): Conv2d(640, 128, kernel_size=(1, 1), stride=(1, 1), bias=False)
(norm2): BatchNorm2d(128, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu2): ReLU(inplace=True)
(conv2): Conv2d(128, 32, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1), bias=False)
)
(denselayer6): _DenseLayer(
(norm1): BatchNorm2d(672, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu1): ReLU(inplace=True)
(conv1): Conv2d(672, 128, kernel_size=(1, 1), stride=(1, 1), bias=False)
(norm2): BatchNorm2d(128, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu2): ReLU(inplace=True)
(conv2): Conv2d(128, 32, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1), bias=False)
)
(denselayer7): _DenseLayer(
(norm1): BatchNorm2d(704, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu1): ReLU(inplace=True)
(conv1): Conv2d(704, 128, kernel_size=(1, 1), stride=(1, 1), bias=False)
(norm2): BatchNorm2d(128, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu2): ReLU(inplace=True)
(conv2): Conv2d(128, 32, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1), bias=False)
)
(denselayer8): _DenseLayer(
(norm1): BatchNorm2d(736, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu1): ReLU(inplace=True)
(conv1): Conv2d(736, 128, kernel_size=(1, 1), stride=(1, 1), bias=False)
(norm2): BatchNorm2d(128, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu2): ReLU(inplace=True)
(conv2): Conv2d(128, 32, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1), bias=False)
)
(denselayer9): _DenseLayer(
(norm1): BatchNorm2d(768, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu1): ReLU(inplace=True)
(conv1): Conv2d(768, 128, kernel_size=(1, 1), stride=(1, 1), bias=False)
(norm2): BatchNorm2d(128, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu2): ReLU(inplace=True)
(conv2): Conv2d(128, 32, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1), bias=False)
)
(denselayer10): _DenseLayer(
(norm1): BatchNorm2d(800, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu1): ReLU(inplace=True)
(conv1): Conv2d(800, 128, kernel_size=(1, 1), stride=(1, 1), bias=False)
(norm2): BatchNorm2d(128, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu2): ReLU(inplace=True)
(conv2): Conv2d(128, 32, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1), bias=False)
)
(denselayer11): _DenseLayer(
(norm1): BatchNorm2d(832, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu1): ReLU(inplace=True)
(conv1): Conv2d(832, 128, kernel_size=(1, 1), stride=(1, 1), bias=False)
(norm2): BatchNorm2d(128, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu2): ReLU(inplace=True)
(conv2): Conv2d(128, 32, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1), bias=False)
)
(denselayer12): _DenseLayer(
(norm1): BatchNorm2d(864, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu1): ReLU(inplace=True)
(conv1): Conv2d(864, 128, kernel_size=(1, 1), stride=(1, 1), bias=False)
(norm2): BatchNorm2d(128, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu2): ReLU(inplace=True)
(conv2): Conv2d(128, 32, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1), bias=False)
)
(denselayer13): _DenseLayer(
(norm1): BatchNorm2d(896, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu1): ReLU(inplace=True)
(conv1): Conv2d(896, 128, kernel_size=(1, 1), stride=(1, 1), bias=False)
(norm2): BatchNorm2d(128, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu2): ReLU(inplace=True)
(conv2): Conv2d(128, 32, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1), bias=False)
)
(denselayer14): _DenseLayer(
(norm1): BatchNorm2d(928, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu1): ReLU(inplace=True)
(conv1): Conv2d(928, 128, kernel_size=(1, 1), stride=(1, 1), bias=False)
(norm2): BatchNorm2d(128, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu2): ReLU(inplace=True)
(conv2): Conv2d(128, 32, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1), bias=False)
)
(denselayer15): _DenseLayer(
(norm1): BatchNorm2d(960, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu1): ReLU(inplace=True)
(conv1): Conv2d(960, 128, kernel_size=(1, 1), stride=(1, 1), bias=False)
(norm2): BatchNorm2d(128, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu2): ReLU(inplace=True)
(conv2): Conv2d(128, 32, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1), bias=False)
)
(denselayer16): _DenseLayer(
(norm1): BatchNorm2d(992, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu1): ReLU(inplace=True)
(conv1): Conv2d(992, 128, kernel_size=(1, 1), stride=(1, 1), bias=False)
(norm2): BatchNorm2d(128, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu2): ReLU(inplace=True)
(conv2): Conv2d(128, 32, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1), bias=False)
)
)
(norm5): BatchNorm2d(1024, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
)
(classifier): Linear(in_features=1024, out_features=6, bias=True)
)
このモデルを使って推論を行います。まず、polyps の画像を 1 枚選び、訓練時と同じ前処理を施します。その後、前処理をした画像をモデルに入力し、予測結果を表示させます。
image_path = 'kvasir/test/polyps/18a31930-8305-49a8-8bb4-1baf35da8c3e.jpg'
image = PIL.Image.open(image_path).convert('RGB')
input_tensor = transform(image).unsqueeze(0).to(device)
with torch.no_grad():
score = model(input_tensor)[0]
output = pd.DataFrame({
'class': labels,
'probability': torch.softmax(score, axis=0).cpu().detach().numpy()
})
output
| class | probability | |
|---|---|---|
| 0 | esophagitis | 0.002280 |
| 1 | normal-cecum | 0.005355 |
| 2 | normal-pylorus | 0.002883 |
| 3 | normal-z-line | 0.002201 |
| 4 | polyps | 0.984380 |
| 5 | ulcerative-colitis | 0.002901 |
次に、別の例を見てみましょう。esophagitis の画像をモデルに入力し、推論を行います。
image_path = 'kvasir/test/esophagitis/ceb61e27-08b3-4887-8bde-3c8f6c537e28.jpg'
image = PIL.Image.open(image_path).convert('RGB')
input_tensor = transform(image).unsqueeze(0).to(device)
with torch.no_grad():
score = model(input_tensor)[0]
pd.DataFrame({
'class': labels,
'probability': torch.softmax(score, axis=0).cpu().detach().numpy()
})
| class | probability | |
|---|---|---|
| 0 | esophagitis | 0.951632 |
| 1 | normal-cecum | 0.002268 |
| 2 | normal-pylorus | 0.010755 |
| 3 | normal-z-line | 0.015851 |
| 4 | polyps | 0.003193 |
| 5 | ulcerative-colitis | 0.016302 |
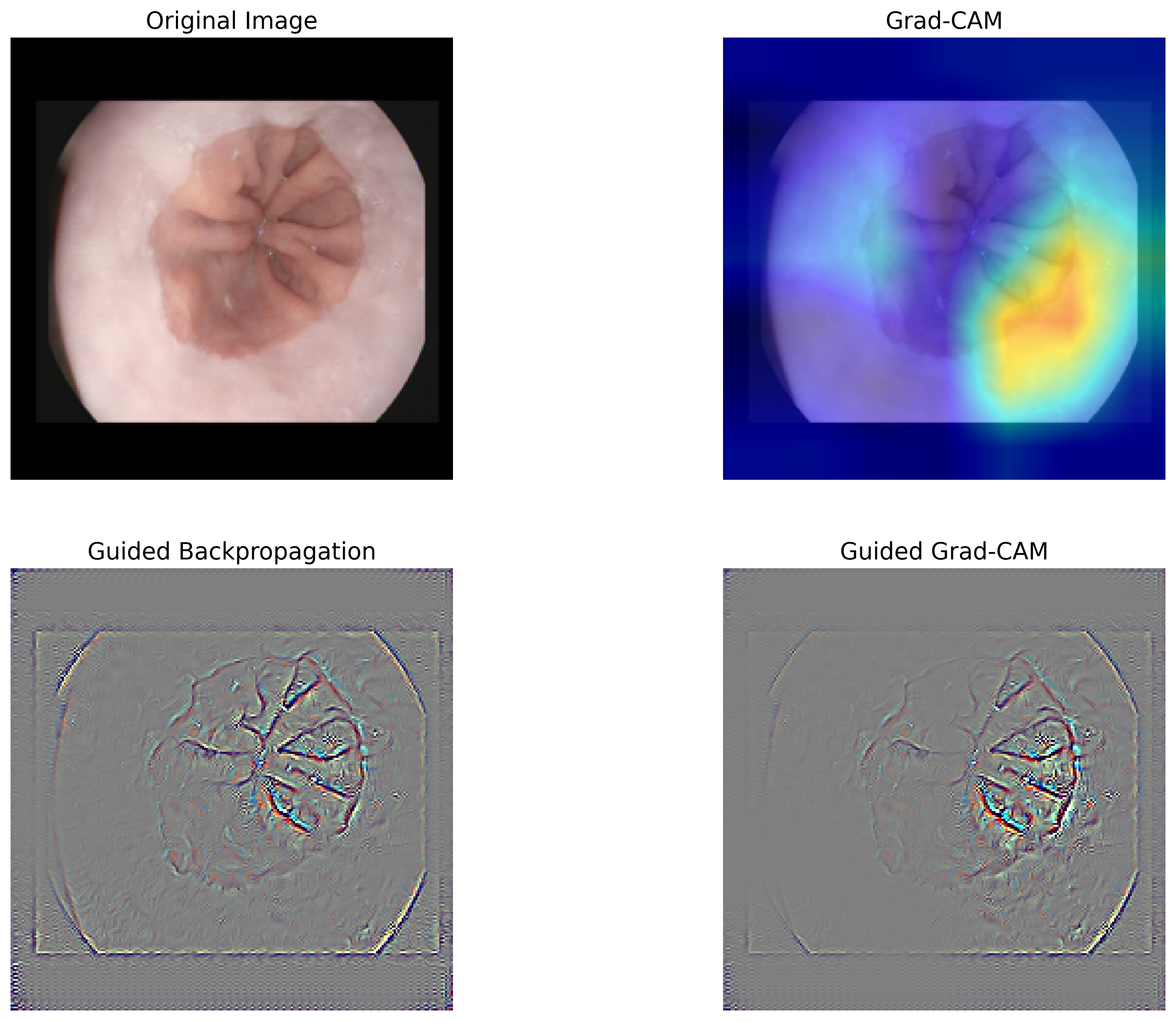
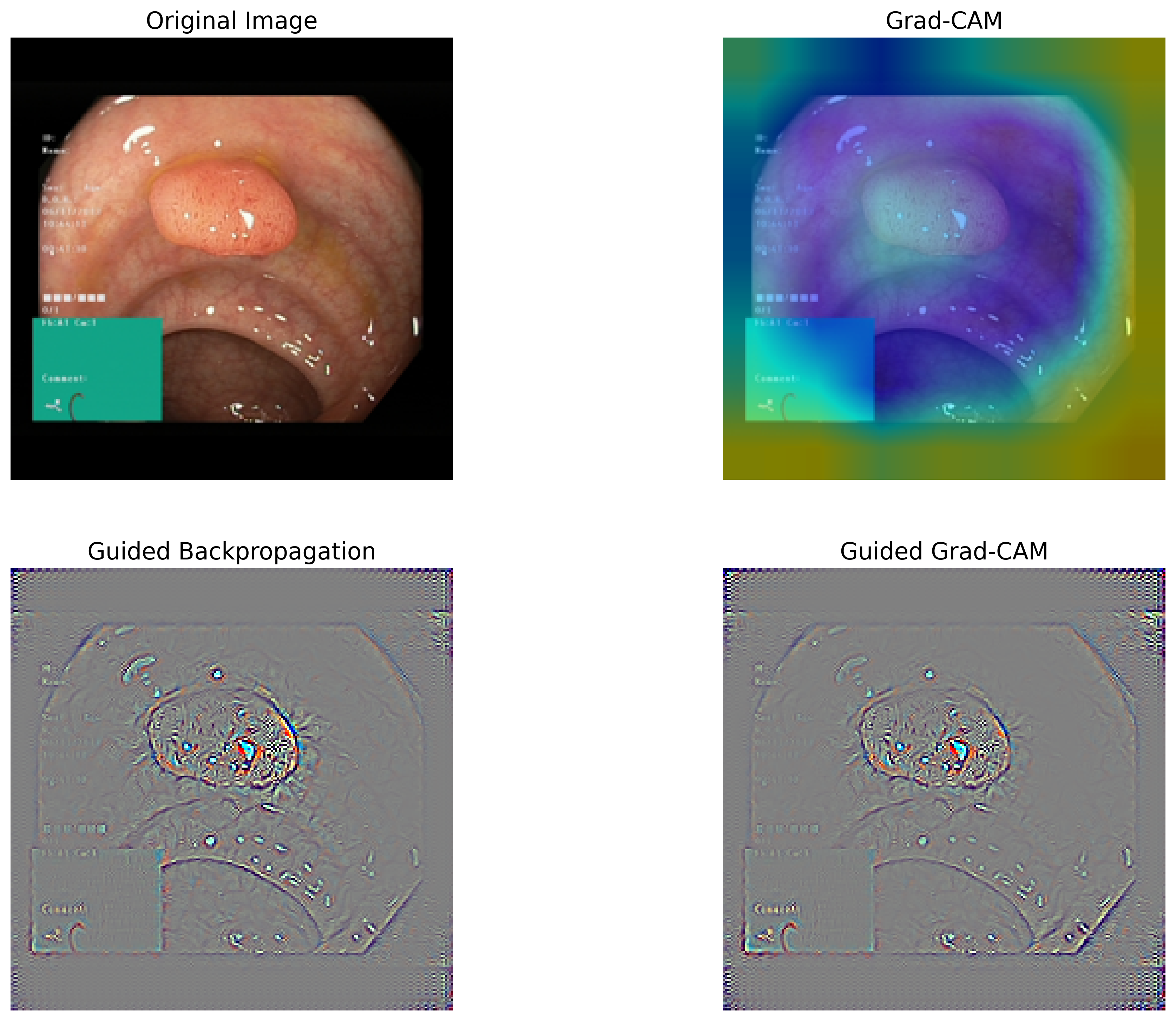
3.4.6. 分類根拠の可視化#
畳み込みニューラルネットワークを用いた画像分類では、畳み込み層で抽出された特徴マップが分類に大きな影響を与えています。そのため、最後の畳み込み層で得られた特徴マップと、それに対応する重みを可視化することで、モデルがどの部分に注目して分類を行ったのか、つまり判断の根拠を明確にすることができます。
本節では、Grad-CAM(Gradient-weighted Class Activation Mapping)および Guided Grad-CAM という手法を用いて、モデルの判断根拠を可視化します。可視化には Python の grad-cam パッケージを使用します。必要に応じてインストールし、grad-cam のチュートリアルを参考にしながら、Grad-CAM および Guided Grad-CAM を計算し、可視化するための関数を定義します。
def viz(image_path):
# load models
labels = ['esophagitis', 'normal-cecum', 'normal-pylorus', 'normal-z-line', 'polyps', 'ulcerative-colitis']
model = torchvision.models.densenet121(weights='DEFAULT')
in_features = model.classifier.in_features
model.classifier = torch.nn.Linear(in_features, 6)
model.load_state_dict(torch.load('kvasir.pth'))
model.to(device)
model.eval()
# load image
image = PIL.Image.open(image_path).convert('RGB')
input_tensor = transform(image).unsqueeze(0).to(device)
rgb_img = cv2.imread(image_path, 1)[:, :, ::-1]
rgb_img = np.float32(np.array(SquareResize()(image))) / 255
# Grad-CAM
with pytorch_grad_cam.GradCAM(model=model, target_layers=[model.features.denseblock4.denselayer16]) as cam:
cam.batch_size = 32
grayscale_cam = cam(input_tensor=input_tensor, targets=None,aug_smooth=True, eigen_smooth=True)
grayscale_cam = grayscale_cam[0, :]
cam_image = pytorch_grad_cam.utils.image.show_cam_on_image(rgb_img, grayscale_cam, use_rgb=True)
prob = torch.softmax(cam.outputs[0], axis=0).cpu().detach().numpy()
gb_model = pytorch_grad_cam.GuidedBackpropReLUModel(model=model, device=device)
gb = gb_model(input_tensor, target_category=None)
cam_mask = np.stack([grayscale_cam, grayscale_cam, grayscale_cam], axis=-1)
cam_gb = pytorch_grad_cam.utils.image.deprocess_image(cam_mask * gb)
gb = pytorch_grad_cam.utils.image.deprocess_image(gb)
# plot
fig, ax = plt.subplots(2, 2)
ax[0, 0].imshow(rgb_img)
ax[0, 0].axis('off')
ax[0, 0].set_title('Original Image', fontsize=16)
ax[0, 1].imshow(cam_image)
ax[0, 1].axis('off')
ax[0, 1].set_title('Grad-CAM', fontsize=16)
ax[1, 0].imshow(gb)
ax[1, 0].axis('off')
ax[1, 0].set_title('Guided Backpropagation', fontsize=16)
ax[1, 1].imshow(cam_gb)
ax[1, 1].axis('off')
ax[1, 1].set_title('Guided Grad-CAM', fontsize=16)
print(pd.DataFrame({'class': labels, 'probability': prob}))
fig.show()
次に、いくつかの画像をこの可視化関数に入力し、モデルの予測結果とその判断根拠を可視化します。これにより、モデルがどの部分に注目して分類を行ったのかを視覚的に確認することができます。必要に応じて、他の画像を入力し、それぞれの分類結果と判断根拠を可視化してみてください。
viz('kvasir/test/polyps/20cb9bd3-af0e-44ea-98a2-186b148d2595.jpg')
class probability
0 esophagitis 0.001494
1 normal-cecum 0.005374
2 normal-pylorus 0.005003
3 normal-z-line 0.001041
4 polyps 0.985794
5 ulcerative-colitis 0.001293
viz('kvasir/test/normal-z-line/96b06b18-6250-484f-955a-6f0179db08a5.jpg')
class probability
0 esophagitis 0.107656
1 normal-cecum 0.001489
2 normal-pylorus 0.003190
3 normal-z-line 0.885581
4 polyps 0.001398
5 ulcerative-colitis 0.000685