5.2. 演習:レントゲン画像歯検出#
歯や歯茎の状態を確認するためにレントゲンを使用することは、歯科検査において欠かせません。もし、レントゲン画像から歯の領域を自動で検出し、その状態、たとえば虫歯の有無を判定できるようになれば、歯科医師の負担を軽減し、診断をより迅速かつ正確に行うことが可能です。このような自動化を実現するには、画像内の歯の領域を正確に分離するセグメンテーション技術が必要です。本節では、レントゲン画像を用いた歯のセグメンテーションの方法を学び、歯科検査を効率化する支援方法を考えていきます。
5.2.1. 演習準備#
5.2.1.1. ライブラリ#
本節で必要なライブラリを読み込みます。os、random、NumPy、Pandas、Matplotlib、Pillow(PIL)は訓練過程の可視化や推論結果の表示に利用します。scikit-image(skimage)はマスクから輪郭線を計算する際に使用します。さらに、torch、torchvision、torchmetrics はインスタンスセグメンテーションモデルの訓練、検証、推論に利用します。
import os
import random
import numpy as np
import pandas as pd
import matplotlib.pyplot as plt
import PIL
import skimage
import torch
import torchvision
import torchvision.transforms.v2
import torchmetrics
print(f'torch v{torch.__version__}; torchvision v{torchvision.__version__}')
torch v2.8.0+cu126; torchvision v0.23.0+cu126
ライブラリの読み込み時に ImportError や ModuleNotFoundError が発生した場合は、該当するライブラリをインストールしてください。ライブラリのバージョンを揃える必要はありませんが、PyTorch(torch)および torchvision が上記のバージョンと異なる時、実行中に警告メッセージが現れたり、同じ結果にならなかったりする可能性があります。
5.2.1.2. データセット#
本章では、Kaggle にて CC0 ライセンスのもとで公開されている Teeth Segmentation on dental X-ray images を使用します。このデータセットは、歯のレントゲン画像からなるデータセットであり、歯を領域をポリゴン囲んだアノテーションが含まれています。アノテーションはポリゴンの座標を記載した数値データと画像として保存されたマスクの両方が用意されています。マスク画像は RGB カラー画像で、画像内の色が各歯の番号に対応しています。例えば、13 番目の歯は色が (R, G, B) = (1, 1, 1)、12 番目の歯は (2, 2, 2) のように、32 番目の歯までそれぞれ異なる色で対応付けられています[1]。歯の番号と色の対応関係は、obj_class_to_machine_color.json ファイルに保存されています。歯の番号を区別して取り扱う際は、この対応表を使用してデータを取得する必要があります。
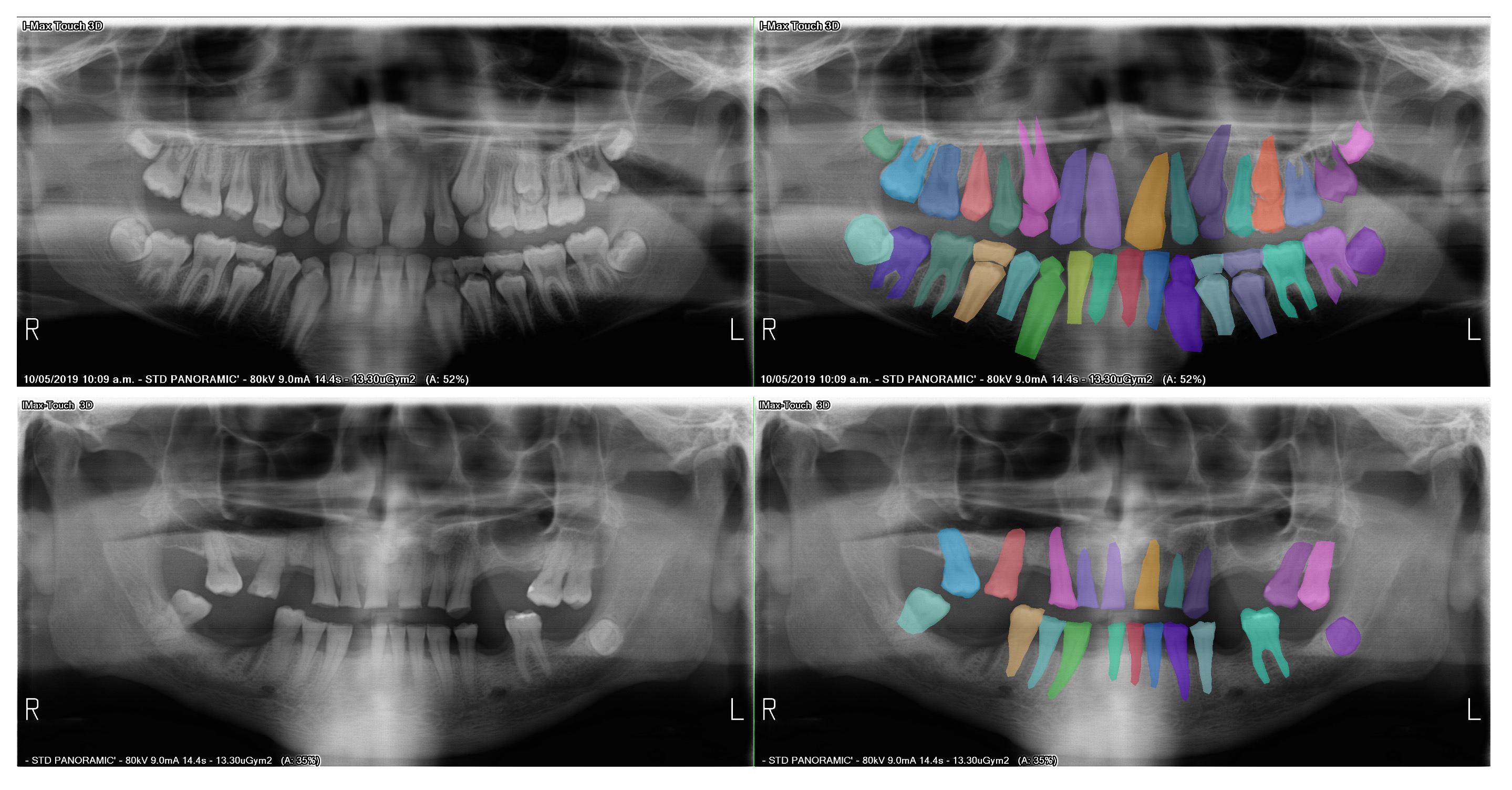
Fig. 5.3 Teeth Segmentation on dental X-ray images データセットのサンプル画像とマスク画像。#
オリジナルのデータセットはやや大きいため、本節では、オリジナルのデータセットから 80 枚の画像を抽出して、そのうち 60 枚を訓練データ、10 枚を検証データ、10 枚を検証データとして整理したものを利用します。Jupyter Notebook では、以下のコマンドを実行することで、データセットをダウンロードできます。
!wget https://dl.biopapyrus.jp/data/teethsegm.zip
!unzip teethsegm.zip
5.2.1.3. 前処理#
本節では、歯の番号を区別せずにインスタンスセグメンテーションを行います。そのために、前述のデータセットをインスタンスセグメンテーションの学習に利用できる形式に変換する処理(TeethDataset)を定義します。
Teeth Segmentation on dental X-ray images データセットに含まれるマスク画像は RGB 画像で、例えば画素値が (1, 1, 1) の部分が 13 番目の歯、(2, 2, 2) の部分が 12 番目の歯に対応しています。TeethDataset では、この RGB 画像を基に、まず画素値が (1, 1, 1) の部分を判定して 1 枚のバイナリマスクを作成し、次に画素値が (2, 2, 2) の部分について同様にバイナリマスクを作成します。この操作を繰り返し、32 本の歯に対応するマスクを生成します((mask == labels[:, None, None]).to(dtype=torch.uint8))。さらに、画像内に該当する歯が存在しない場合、その歯に対応するマスクの画素値はすべて 0 になります。このような無効なマスクを削除する処理(masks[has_tooth])も実装しています。最後に、生成したマスクを PyTorch で扱える形式に変換する処理を行います。
class TeethDataset(torch.utils.data.Dataset):
def __init__(self, root):
self.root = root
self.images, self.masks = self.__load_datasets(self.root)
self.transforms = torchvision.transforms.v2.Compose([
torchvision.transforms.v2.ToDtype(torch.float, scale=True),
torchvision.transforms.v2.ToPureTensor()
])
def __getitem__(self, idx):
image = torchvision.io.read_image(self.images[idx])
mask = torchvision.io.read_image(self.masks[idx])
# create labels, masks, bboxes for training from the original mask
labels = torch.tensor([_ for _ in range(1, 33)])
masks = (mask == labels[:, None, None]).to(dtype=torch.uint8)
has_tooth = [_.sum() > 0 for _ in masks]
labels = labels[has_tooth]
masks = masks[has_tooth]
boxes = torchvision.ops.boxes.masks_to_boxes(masks)
# convert teeth number to 1 (ignore the teeth number)
labels = torch.ones((len(labels), ), dtype=torch.int64)
# format image and annotation for training
image = torchvision.tv_tensors.Image(image)
target = {
'boxes': torchvision.tv_tensors.BoundingBoxes(boxes, format='XYXY', canvas_size=torchvision.transforms.v2.functional.get_size(image)),
'masks': torchvision.tv_tensors.Mask(masks),
'labels': labels,
}
image, target = self.transforms(image, target)
return image, target
def __len__(self):
return len(self.images)
def __load_datasets(self, root):
images = []
masks = []
for image_fpath in os.listdir(os.path.join(root, 'images')):
image_fpath = os.path.join(root, 'images', image_fpath)
if os.path.splitext(image_fpath)[1] == '.jpg':
image_fname = os.path.basename(image_fpath)
mask_fpath = os.path.join(root, 'masks', os.path.splitext(image_fname)[0] + '.png')
if os.path.exists(mask_fpath):
images.append(image_fpath)
masks.append(mask_fpath)
return images, masks
通常、モデルの訓練では、データ拡張として画像の拡大縮小、平行移動、回転などを適用することが一般的です。しかし、歯のレントゲン画像の場合、過度なデータ拡張を行うと、本来の画像情報から逸脱し、モデルの学習に悪影響を及ぼす可能性があります。そのため、適切なデータ拡張手法を慎重に選択することが重要です。
5.2.1.4. 計算デバイス#
PyTorch が GPU を認識できる場合は GPU を利用し、認識できない場合は CPU を利用するように計算デバイスを設定します。
device = torch.device('cuda') if torch.cuda.is_available() else torch.device('cpu')
5.2.2. モデル構築#
Mask R-CNN は高性能なインスタンスセグメンテーションを行うアーキテクチャで、torchvision.models モジュールに含まれています。しかし、torchvision.models で提供されている Mask R-CNN は、COCO データセット向けに設計されており、車や人などの 90 種類の一般的なオブジェクトを対象としています。そのため、そのままでは歯のインスタンスセグメンテーションに適用することができません。
歯のセグメンテーションに対応させるためには、Mask R-CNN の分類モジュールの出力層のユニット数を修正する必要があります。この修正はモデルを呼び出すたびに行う必要があり、手間がかかります。そこで、指定したクラス数に応じてアーキテクチャを生成し、必要に応じて修正を加えられるように、一連の処理を関数として定義します。なお、インスタンスセグメンテーションは物体検出と同様に背景を 1 つのクラスとして扱うため、出力層の数を修正する際には、検出対象のクラス数に 1 を加えた数にする必要があります。
def maskrcnn(num_classes, weights=None):
num_classes = num_classes + 1
model = torchvision.models.detection.maskrcnn_resnet50_fpn(weights='DEFAULT')
in_features = model.roi_heads.box_predictor.cls_score.in_features
model.roi_heads.box_predictor = torchvision.models.detection.faster_rcnn.FastRCNNPredictor(in_features, num_classes)
in_features_mask = model.roi_heads.mask_predictor.conv5_mask.in_channels
model.roi_heads.mask_predictor = torchvision.models.detection.mask_rcnn.MaskRCNNPredictor(in_features_mask, 256, num_classes)
if weights is not None:
model.load_state_dict(torch.load(weights))
return model
model = maskrcnn(num_classes=1)
model.to(device)
Show code cell output
MaskRCNN(
(transform): GeneralizedRCNNTransform(
Normalize(mean=[0.485, 0.456, 0.406], std=[0.229, 0.224, 0.225])
Resize(min_size=(800,), max_size=1333, mode='bilinear')
)
(backbone): BackboneWithFPN(
(body): IntermediateLayerGetter(
(conv1): Conv2d(3, 64, kernel_size=(7, 7), stride=(2, 2), padding=(3, 3), bias=False)
(bn1): FrozenBatchNorm2d(64, eps=0.0)
(relu): ReLU(inplace=True)
(maxpool): MaxPool2d(kernel_size=3, stride=2, padding=1, dilation=1, ceil_mode=False)
(layer1): Sequential(
(0): Bottleneck(
(conv1): Conv2d(64, 64, kernel_size=(1, 1), stride=(1, 1), bias=False)
(bn1): FrozenBatchNorm2d(64, eps=0.0)
(conv2): Conv2d(64, 64, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1), bias=False)
(bn2): FrozenBatchNorm2d(64, eps=0.0)
(conv3): Conv2d(64, 256, kernel_size=(1, 1), stride=(1, 1), bias=False)
(bn3): FrozenBatchNorm2d(256, eps=0.0)
(relu): ReLU(inplace=True)
(downsample): Sequential(
(0): Conv2d(64, 256, kernel_size=(1, 1), stride=(1, 1), bias=False)
(1): FrozenBatchNorm2d(256, eps=0.0)
)
)
(1): Bottleneck(
(conv1): Conv2d(256, 64, kernel_size=(1, 1), stride=(1, 1), bias=False)
(bn1): FrozenBatchNorm2d(64, eps=0.0)
(conv2): Conv2d(64, 64, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1), bias=False)
(bn2): FrozenBatchNorm2d(64, eps=0.0)
(conv3): Conv2d(64, 256, kernel_size=(1, 1), stride=(1, 1), bias=False)
(bn3): FrozenBatchNorm2d(256, eps=0.0)
(relu): ReLU(inplace=True)
)
(2): Bottleneck(
(conv1): Conv2d(256, 64, kernel_size=(1, 1), stride=(1, 1), bias=False)
(bn1): FrozenBatchNorm2d(64, eps=0.0)
(conv2): Conv2d(64, 64, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1), bias=False)
(bn2): FrozenBatchNorm2d(64, eps=0.0)
(conv3): Conv2d(64, 256, kernel_size=(1, 1), stride=(1, 1), bias=False)
(bn3): FrozenBatchNorm2d(256, eps=0.0)
(relu): ReLU(inplace=True)
)
)
(layer2): Sequential(
(0): Bottleneck(
(conv1): Conv2d(256, 128, kernel_size=(1, 1), stride=(1, 1), bias=False)
(bn1): FrozenBatchNorm2d(128, eps=0.0)
(conv2): Conv2d(128, 128, kernel_size=(3, 3), stride=(2, 2), padding=(1, 1), bias=False)
(bn2): FrozenBatchNorm2d(128, eps=0.0)
(conv3): Conv2d(128, 512, kernel_size=(1, 1), stride=(1, 1), bias=False)
(bn3): FrozenBatchNorm2d(512, eps=0.0)
(relu): ReLU(inplace=True)
(downsample): Sequential(
(0): Conv2d(256, 512, kernel_size=(1, 1), stride=(2, 2), bias=False)
(1): FrozenBatchNorm2d(512, eps=0.0)
)
)
(1): Bottleneck(
(conv1): Conv2d(512, 128, kernel_size=(1, 1), stride=(1, 1), bias=False)
(bn1): FrozenBatchNorm2d(128, eps=0.0)
(conv2): Conv2d(128, 128, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1), bias=False)
(bn2): FrozenBatchNorm2d(128, eps=0.0)
(conv3): Conv2d(128, 512, kernel_size=(1, 1), stride=(1, 1), bias=False)
(bn3): FrozenBatchNorm2d(512, eps=0.0)
(relu): ReLU(inplace=True)
)
(2): Bottleneck(
(conv1): Conv2d(512, 128, kernel_size=(1, 1), stride=(1, 1), bias=False)
(bn1): FrozenBatchNorm2d(128, eps=0.0)
(conv2): Conv2d(128, 128, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1), bias=False)
(bn2): FrozenBatchNorm2d(128, eps=0.0)
(conv3): Conv2d(128, 512, kernel_size=(1, 1), stride=(1, 1), bias=False)
(bn3): FrozenBatchNorm2d(512, eps=0.0)
(relu): ReLU(inplace=True)
)
(3): Bottleneck(
(conv1): Conv2d(512, 128, kernel_size=(1, 1), stride=(1, 1), bias=False)
(bn1): FrozenBatchNorm2d(128, eps=0.0)
(conv2): Conv2d(128, 128, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1), bias=False)
(bn2): FrozenBatchNorm2d(128, eps=0.0)
(conv3): Conv2d(128, 512, kernel_size=(1, 1), stride=(1, 1), bias=False)
(bn3): FrozenBatchNorm2d(512, eps=0.0)
(relu): ReLU(inplace=True)
)
)
(layer3): Sequential(
(0): Bottleneck(
(conv1): Conv2d(512, 256, kernel_size=(1, 1), stride=(1, 1), bias=False)
(bn1): FrozenBatchNorm2d(256, eps=0.0)
(conv2): Conv2d(256, 256, kernel_size=(3, 3), stride=(2, 2), padding=(1, 1), bias=False)
(bn2): FrozenBatchNorm2d(256, eps=0.0)
(conv3): Conv2d(256, 1024, kernel_size=(1, 1), stride=(1, 1), bias=False)
(bn3): FrozenBatchNorm2d(1024, eps=0.0)
(relu): ReLU(inplace=True)
(downsample): Sequential(
(0): Conv2d(512, 1024, kernel_size=(1, 1), stride=(2, 2), bias=False)
(1): FrozenBatchNorm2d(1024, eps=0.0)
)
)
(1): Bottleneck(
(conv1): Conv2d(1024, 256, kernel_size=(1, 1), stride=(1, 1), bias=False)
(bn1): FrozenBatchNorm2d(256, eps=0.0)
(conv2): Conv2d(256, 256, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1), bias=False)
(bn2): FrozenBatchNorm2d(256, eps=0.0)
(conv3): Conv2d(256, 1024, kernel_size=(1, 1), stride=(1, 1), bias=False)
(bn3): FrozenBatchNorm2d(1024, eps=0.0)
(relu): ReLU(inplace=True)
)
(2): Bottleneck(
(conv1): Conv2d(1024, 256, kernel_size=(1, 1), stride=(1, 1), bias=False)
(bn1): FrozenBatchNorm2d(256, eps=0.0)
(conv2): Conv2d(256, 256, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1), bias=False)
(bn2): FrozenBatchNorm2d(256, eps=0.0)
(conv3): Conv2d(256, 1024, kernel_size=(1, 1), stride=(1, 1), bias=False)
(bn3): FrozenBatchNorm2d(1024, eps=0.0)
(relu): ReLU(inplace=True)
)
(3): Bottleneck(
(conv1): Conv2d(1024, 256, kernel_size=(1, 1), stride=(1, 1), bias=False)
(bn1): FrozenBatchNorm2d(256, eps=0.0)
(conv2): Conv2d(256, 256, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1), bias=False)
(bn2): FrozenBatchNorm2d(256, eps=0.0)
(conv3): Conv2d(256, 1024, kernel_size=(1, 1), stride=(1, 1), bias=False)
(bn3): FrozenBatchNorm2d(1024, eps=0.0)
(relu): ReLU(inplace=True)
)
(4): Bottleneck(
(conv1): Conv2d(1024, 256, kernel_size=(1, 1), stride=(1, 1), bias=False)
(bn1): FrozenBatchNorm2d(256, eps=0.0)
(conv2): Conv2d(256, 256, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1), bias=False)
(bn2): FrozenBatchNorm2d(256, eps=0.0)
(conv3): Conv2d(256, 1024, kernel_size=(1, 1), stride=(1, 1), bias=False)
(bn3): FrozenBatchNorm2d(1024, eps=0.0)
(relu): ReLU(inplace=True)
)
(5): Bottleneck(
(conv1): Conv2d(1024, 256, kernel_size=(1, 1), stride=(1, 1), bias=False)
(bn1): FrozenBatchNorm2d(256, eps=0.0)
(conv2): Conv2d(256, 256, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1), bias=False)
(bn2): FrozenBatchNorm2d(256, eps=0.0)
(conv3): Conv2d(256, 1024, kernel_size=(1, 1), stride=(1, 1), bias=False)
(bn3): FrozenBatchNorm2d(1024, eps=0.0)
(relu): ReLU(inplace=True)
)
)
(layer4): Sequential(
(0): Bottleneck(
(conv1): Conv2d(1024, 512, kernel_size=(1, 1), stride=(1, 1), bias=False)
(bn1): FrozenBatchNorm2d(512, eps=0.0)
(conv2): Conv2d(512, 512, kernel_size=(3, 3), stride=(2, 2), padding=(1, 1), bias=False)
(bn2): FrozenBatchNorm2d(512, eps=0.0)
(conv3): Conv2d(512, 2048, kernel_size=(1, 1), stride=(1, 1), bias=False)
(bn3): FrozenBatchNorm2d(2048, eps=0.0)
(relu): ReLU(inplace=True)
(downsample): Sequential(
(0): Conv2d(1024, 2048, kernel_size=(1, 1), stride=(2, 2), bias=False)
(1): FrozenBatchNorm2d(2048, eps=0.0)
)
)
(1): Bottleneck(
(conv1): Conv2d(2048, 512, kernel_size=(1, 1), stride=(1, 1), bias=False)
(bn1): FrozenBatchNorm2d(512, eps=0.0)
(conv2): Conv2d(512, 512, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1), bias=False)
(bn2): FrozenBatchNorm2d(512, eps=0.0)
(conv3): Conv2d(512, 2048, kernel_size=(1, 1), stride=(1, 1), bias=False)
(bn3): FrozenBatchNorm2d(2048, eps=0.0)
(relu): ReLU(inplace=True)
)
(2): Bottleneck(
(conv1): Conv2d(2048, 512, kernel_size=(1, 1), stride=(1, 1), bias=False)
(bn1): FrozenBatchNorm2d(512, eps=0.0)
(conv2): Conv2d(512, 512, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1), bias=False)
(bn2): FrozenBatchNorm2d(512, eps=0.0)
(conv3): Conv2d(512, 2048, kernel_size=(1, 1), stride=(1, 1), bias=False)
(bn3): FrozenBatchNorm2d(2048, eps=0.0)
(relu): ReLU(inplace=True)
)
)
)
(fpn): FeaturePyramidNetwork(
(inner_blocks): ModuleList(
(0): Conv2dNormActivation(
(0): Conv2d(256, 256, kernel_size=(1, 1), stride=(1, 1))
)
(1): Conv2dNormActivation(
(0): Conv2d(512, 256, kernel_size=(1, 1), stride=(1, 1))
)
(2): Conv2dNormActivation(
(0): Conv2d(1024, 256, kernel_size=(1, 1), stride=(1, 1))
)
(3): Conv2dNormActivation(
(0): Conv2d(2048, 256, kernel_size=(1, 1), stride=(1, 1))
)
)
(layer_blocks): ModuleList(
(0-3): 4 x Conv2dNormActivation(
(0): Conv2d(256, 256, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))
)
)
(extra_blocks): LastLevelMaxPool()
)
)
(rpn): RegionProposalNetwork(
(anchor_generator): AnchorGenerator()
(head): RPNHead(
(conv): Sequential(
(0): Conv2dNormActivation(
(0): Conv2d(256, 256, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))
(1): ReLU(inplace=True)
)
)
(cls_logits): Conv2d(256, 3, kernel_size=(1, 1), stride=(1, 1))
(bbox_pred): Conv2d(256, 12, kernel_size=(1, 1), stride=(1, 1))
)
)
(roi_heads): RoIHeads(
(box_roi_pool): MultiScaleRoIAlign(featmap_names=['0', '1', '2', '3'], output_size=(7, 7), sampling_ratio=2)
(box_head): TwoMLPHead(
(fc6): Linear(in_features=12544, out_features=1024, bias=True)
(fc7): Linear(in_features=1024, out_features=1024, bias=True)
)
(box_predictor): FastRCNNPredictor(
(cls_score): Linear(in_features=1024, out_features=2, bias=True)
(bbox_pred): Linear(in_features=1024, out_features=8, bias=True)
)
(mask_roi_pool): MultiScaleRoIAlign(featmap_names=['0', '1', '2', '3'], output_size=(14, 14), sampling_ratio=2)
(mask_head): MaskRCNNHeads(
(0): Conv2dNormActivation(
(0): Conv2d(256, 256, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))
(1): ReLU(inplace=True)
)
(1): Conv2dNormActivation(
(0): Conv2d(256, 256, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))
(1): ReLU(inplace=True)
)
(2): Conv2dNormActivation(
(0): Conv2d(256, 256, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))
(1): ReLU(inplace=True)
)
(3): Conv2dNormActivation(
(0): Conv2d(256, 256, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))
(1): ReLU(inplace=True)
)
)
(mask_predictor): MaskRCNNPredictor(
(conv5_mask): ConvTranspose2d(256, 256, kernel_size=(2, 2), stride=(2, 2))
(relu): ReLU(inplace=True)
(mask_fcn_logits): Conv2d(256, 2, kernel_size=(1, 1), stride=(1, 1))
)
)
)
5.2.3. モデル訓練#
モデルが学習データを効率的に学習できるように、学習アルゴリズム(optimizer)、学習率(lr)、および学習率を調整するスケジューラ(lr_scheduler)を設定します。なお、インスタンスセグメンテーションでは、すべてのピクセルに対して予測結果(分類結果)とラベルの誤差を計算する損失関数を定義する必要がありますが、この損失関数はすでにモデル内で定義されているため、ここで新たに定義する必要はありません。
optimizer = torch.optim.AdamW(model.parameters(), lr=1e-4, weight_decay=1e-4)
lr_scheduler = torch.optim.lr_scheduler.StepLR(optimizer, step_size=3, gamma=0.1)
次に、訓練サブセットおよび検証サブセットを読み込み、モデルに入力できる形式に整えます。
train_loader = torch.utils.data.DataLoader(
TeethDataset('teethsegm/train'),
batch_size=4, shuffle=True, collate_fn=lambda x: tuple(zip(*x)))
valid_loader = torch.utils.data.DataLoader(
TeethDataset('teethsegm/valid'),
batch_size=4, shuffle=False, collate_fn=lambda x: tuple(zip(*x)))
準備が整ったら、訓練を開始します。訓練プロセスでは、訓練と検証を交互に繰り返します。訓練では、訓練データを使ってモデルのパラメータを更新し、その際の損失(誤差)を記録します。検証では、検証データを使ってモデルの予測性能(mAP や IoU)を計算し、その結果を記録します。
num_epochs = 10
metric_dict = []
for epoch in range(num_epochs):
# training phase
model.train()
epoch_loss_dict = {}
for images, targets in train_loader:
images = [img.to(device) for img in images]
targets = [{k: v.to(device) for k, v in t.items()} for t in targets]
batch_loss_dict = model(images, targets)
batch_tol_loss = 0
for loss_type, loss_val in batch_loss_dict.items():
batch_tol_loss += loss_val
if loss_type in epoch_loss_dict:
epoch_loss_dict[f'train_{loss_type}'] += loss_val.item()
else:
epoch_loss_dict[f'train_{loss_type}'] = loss_val.item()
# update weights
optimizer.zero_grad()
batch_tol_loss.backward()
optimizer.step()
lr_scheduler.step()
# validation phase
model.eval()
metric = torchmetrics.detection.mean_ap.MeanAveragePrecision()
iou = 0
n_targets = 0
with torch.no_grad():
for images, targets in valid_loader:
images = [img.to(device) for img in images]
targets = [{k: v.to(device) for k, v in t.items()} for t in targets]
pred_targets = model(images)
metric.update(pred_targets, targets)
for i in range(len(targets)):
pred_mask = pred_targets[i]['masks'].squeeze(1).any(dim=0)
true_mask = targets[i]['masks'].any(dim=0)
iou += torchmetrics.functional.jaccard_index(pred_mask.unsqueeze(0), true_mask.unsqueeze(0), num_classes=1, task='binary')
n_targets += 1
# record training loss
epoch_loss_dict['train_total_loss'] = sum(epoch_loss_dict.values())
metric_dict.append({k: v / len(train_loader) for k, v in epoch_loss_dict.items()})
for k, v in metric.compute().items():
if k != 'classes':
metric_dict[-1][k] = v.item()
metric_dict[-1]['avg_iou'] = iou.item() / n_targets
metric_dict[-1]['epoch'] = epoch + 1
print(metric_dict[-1])
Show code cell output
{'train_loss_classifier': 0.011479345957438152, 'train_loss_box_reg': 0.03140854835510254, 'train_loss_mask': 0.01937209963798523, 'train_loss_objectness': 0.00249134103457133, 'train_loss_rpn_box_reg': 0.003991664946079254, 'train_total_loss': 0.06874299993117651, 'map': 0.5010296702384949, 'map_50': 0.9746415019035339, 'map_75': 0.4743480086326599, 'map_small': -1.0, 'map_medium': 0.2945922017097473, 'map_large': 0.5136563777923584, 'mar_1': 0.020817844197154045, 'mar_10': 0.2275092899799347, 'mar_100': 0.570260226726532, 'mar_small': -1.0, 'mar_medium': 0.4058823585510254, 'mar_large': 0.5813491940498352, 'map_per_class': -1.0, 'mar_100_per_class': -1.0, 'avg_iou': 0.4299659252166748, 'epoch': 1}
{'train_loss_classifier': 0.008575915296872457, 'train_loss_box_reg': 0.022289788722991942, 'train_loss_mask': 0.01755560040473938, 'train_loss_objectness': 0.00027365656569600105, 'train_loss_rpn_box_reg': 0.002937895804643631, 'train_total_loss': 0.051632856794943414, 'map': 0.5697304010391235, 'map_50': 0.9816696047782898, 'map_75': 0.5916833877563477, 'map_small': -1.0, 'map_medium': 0.4229339063167572, 'map_large': 0.579137921333313, 'mar_1': 0.025278810411691666, 'mar_10': 0.26319703459739685, 'mar_100': 0.6397769451141357, 'mar_small': -1.0, 'mar_medium': 0.5117647051811218, 'mar_large': 0.6484127044677734, 'map_per_class': -1.0, 'mar_100_per_class': -1.0, 'avg_iou': 0.5545343399047852, 'epoch': 2}
{'train_loss_classifier': 0.012302353978157043, 'train_loss_box_reg': 0.025302062431971233, 'train_loss_mask': 0.016267017523447672, 'train_loss_objectness': 0.0011291346202294032, 'train_loss_rpn_box_reg': 0.0022896779080231982, 'train_total_loss': 0.05729024646182855, 'map': 0.5723249316215515, 'map_50': 0.9854661822319031, 'map_75': 0.5852181911468506, 'map_small': -1.0, 'map_medium': 0.423562228679657, 'map_large': 0.5779514312744141, 'mar_1': 0.02750929445028305, 'mar_10': 0.26542750000953674, 'mar_100': 0.6423791646957397, 'mar_small': -1.0, 'mar_medium': 0.5529412031173706, 'mar_large': 0.6484127044677734, 'map_per_class': -1.0, 'mar_100_per_class': -1.0, 'avg_iou': 0.6228688716888428, 'epoch': 3}
{'train_loss_classifier': 0.009580006202061972, 'train_loss_box_reg': 0.023113731543223062, 'train_loss_mask': 0.017496782541275024, 'train_loss_objectness': 0.00027786670252680776, 'train_loss_rpn_box_reg': 0.0028051485617955527, 'train_total_loss': 0.05327353555088242, 'map': 0.6246872544288635, 'map_50': 0.987676203250885, 'map_75': 0.7476171851158142, 'map_small': -1.0, 'map_medium': 0.4803968071937561, 'map_large': 0.6335659027099609, 'mar_1': 0.026765799149870872, 'mar_10': 0.27657991647720337, 'mar_100': 0.6869888305664062, 'mar_small': -1.0, 'mar_medium': 0.5588235259056091, 'mar_large': 0.6956349015235901, 'map_per_class': -1.0, 'mar_100_per_class': -1.0, 'avg_iou': 0.6128114700317383, 'epoch': 4}
{'train_loss_classifier': 0.00847939153512319, 'train_loss_box_reg': 0.0202574094136556, 'train_loss_mask': 0.017841337124506633, 'train_loss_objectness': 0.00027715135365724564, 'train_loss_rpn_box_reg': 0.0028409595290819806, 'train_total_loss': 0.04969624895602465, 'map': 0.6225438714027405, 'map_50': 0.9877026677131653, 'map_75': 0.7465952634811401, 'map_small': -1.0, 'map_medium': 0.4661967158317566, 'map_large': 0.6312015056610107, 'mar_1': 0.02490706369280815, 'mar_10': 0.2754646837711334, 'mar_100': 0.6877323389053345, 'mar_small': -1.0, 'mar_medium': 0.5588235259056091, 'mar_large': 0.6964285969734192, 'map_per_class': -1.0, 'mar_100_per_class': -1.0, 'avg_iou': 0.6286535263061523, 'epoch': 5}
{'train_loss_classifier': 0.008252489070097605, 'train_loss_box_reg': 0.018065027395884194, 'train_loss_mask': 0.013969463109970093, 'train_loss_objectness': 6.834429999192556e-05, 'train_loss_rpn_box_reg': 0.0017130342622598013, 'train_total_loss': 0.04206835813820362, 'map': 0.6262150406837463, 'map_50': 0.9878362417221069, 'map_75': 0.7463611960411072, 'map_small': -1.0, 'map_medium': 0.4871683120727539, 'map_large': 0.6359767317771912, 'mar_1': 0.025278810411691666, 'mar_10': 0.2821561396121979, 'mar_100': 0.6899628043174744, 'mar_small': -1.0, 'mar_medium': 0.529411792755127, 'mar_large': 0.7007936239242554, 'map_per_class': -1.0, 'mar_100_per_class': -1.0, 'avg_iou': 0.6258145332336426, 'epoch': 6}
{'train_loss_classifier': 0.007895846168200176, 'train_loss_box_reg': 0.016363452871640524, 'train_loss_mask': 0.014645089705785116, 'train_loss_objectness': 0.00017125693460305532, 'train_loss_rpn_box_reg': 0.0018430655201276144, 'train_total_loss': 0.040918711200356485, 'map': 0.6276139616966248, 'map_50': 0.9878007173538208, 'map_75': 0.7398622632026672, 'map_small': -1.0, 'map_medium': 0.49505844712257385, 'map_large': 0.6364915370941162, 'mar_1': 0.02490706369280815, 'mar_10': 0.28141263127326965, 'mar_100': 0.6910780668258667, 'mar_small': -1.0, 'mar_medium': 0.5411764979362488, 'mar_large': 0.7011904716491699, 'map_per_class': -1.0, 'mar_100_per_class': -1.0, 'avg_iou': 0.6261999607086182, 'epoch': 7}
{'train_loss_classifier': 0.007680356999238332, 'train_loss_box_reg': 0.017280463377634683, 'train_loss_mask': 0.015475576122601828, 'train_loss_objectness': 0.00026905257254838943, 'train_loss_rpn_box_reg': 0.0023739129304885865, 'train_total_loss': 0.04307936200251182, 'map': 0.6300591826438904, 'map_50': 0.9878469109535217, 'map_75': 0.7521981000900269, 'map_small': -1.0, 'map_medium': 0.5104413628578186, 'map_large': 0.6388262510299683, 'mar_1': 0.024535315111279488, 'mar_10': 0.2832713723182678, 'mar_100': 0.6933085322380066, 'mar_small': -1.0, 'mar_medium': 0.5529412031173706, 'mar_large': 0.7027778029441833, 'map_per_class': -1.0, 'mar_100_per_class': -1.0, 'avg_iou': 0.6273415088653564, 'epoch': 8}
{'train_loss_classifier': 0.008191153407096863, 'train_loss_box_reg': 0.019140756130218504, 'train_loss_mask': 0.016015393535296123, 'train_loss_objectness': 0.0003745509621997674, 'train_loss_rpn_box_reg': 0.0028148382902145387, 'train_total_loss': 0.046536692325025796, 'map': 0.629723310470581, 'map_50': 0.9878469109535217, 'map_75': 0.7504862546920776, 'map_small': -1.0, 'map_medium': 0.5104413628578186, 'map_large': 0.6384953856468201, 'mar_1': 0.024535315111279488, 'mar_10': 0.282527893781662, 'mar_100': 0.6918215751647949, 'mar_small': -1.0, 'mar_medium': 0.5529412031173706, 'mar_large': 0.7011904716491699, 'map_per_class': -1.0, 'mar_100_per_class': -1.0, 'avg_iou': 0.6291441917419434, 'epoch': 9}
{'train_loss_classifier': 0.007440189520517985, 'train_loss_box_reg': 0.01594864626725515, 'train_loss_mask': 0.013907363017400106, 'train_loss_objectness': 8.795432125528654e-05, 'train_loss_rpn_box_reg': 0.0016577338178952535, 'train_total_loss': 0.03904188694432378, 'map': 0.6297749280929565, 'map_50': 0.9878469109535217, 'map_75': 0.7505040764808655, 'map_small': -1.0, 'map_medium': 0.5104413628578186, 'map_large': 0.6385549306869507, 'mar_1': 0.024535315111279488, 'mar_10': 0.282527893781662, 'mar_100': 0.6918215751647949, 'mar_small': -1.0, 'mar_medium': 0.5529412031173706, 'mar_large': 0.7011904716491699, 'map_per_class': -1.0, 'mar_100_per_class': -1.0, 'avg_iou': 0.6292304039001465, 'epoch': 10}
訓練データに対する損失と検証データに対する予測性能(mAP)を可視化し、訓練過程を評価します。
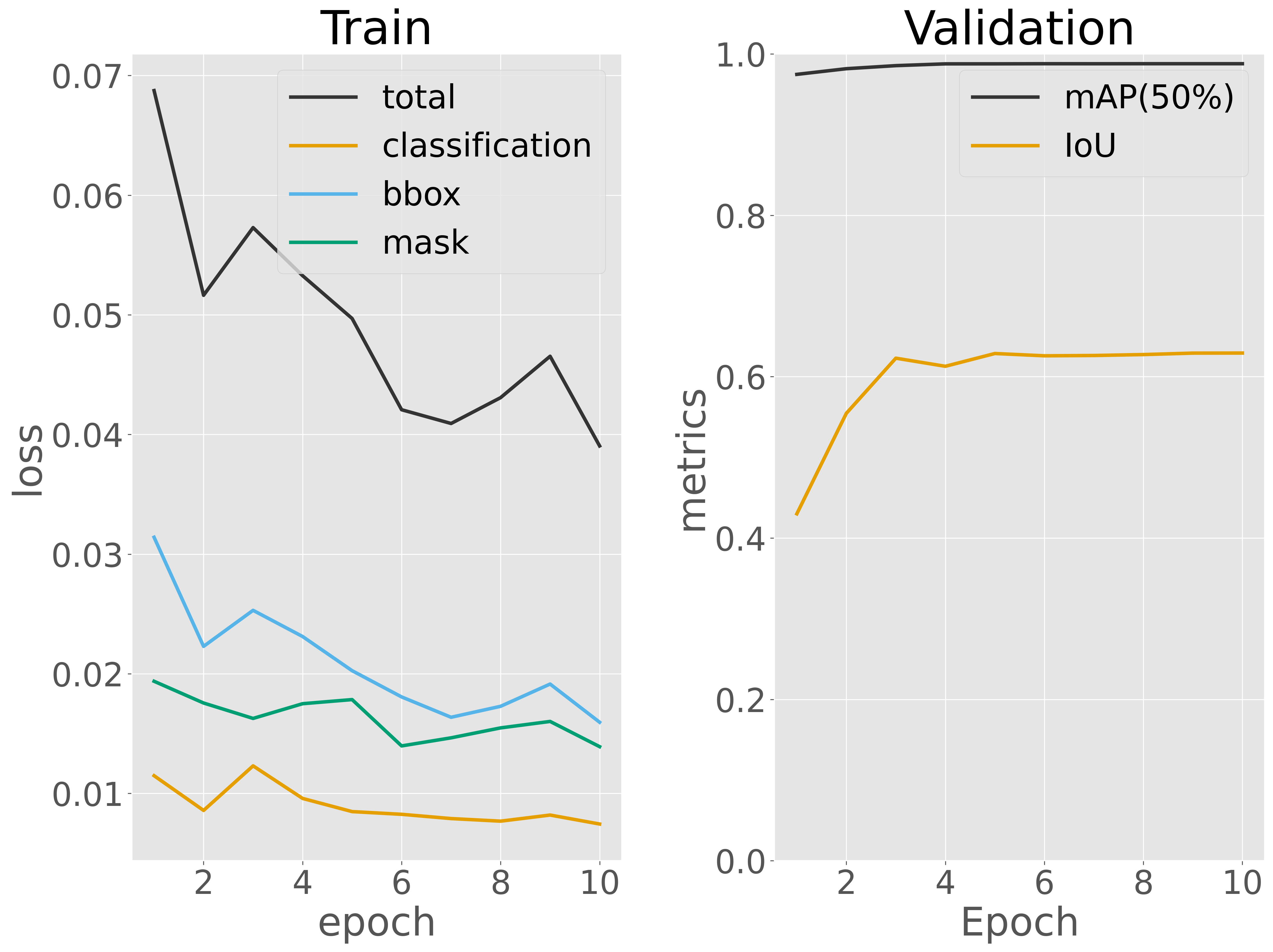
可視化の結果から、エポック数が増えるにつれて訓練データに対する損失が継続的に減少し、5 エポック目から収束し始める傾向がみられました。一方で、検証データに対する検証性能(mAP および IoU)は最初の数エポックですでに高い値に達しいることがわかりました。訓練ではこれで十分と考えらえるので次のステップに進みます。
次に、この手順を他の深層ニューラルネットワークのアーキテクチャ(例えば U-Net など)に適用し、それぞれの検証性能を比較します。この比較により、データセットに最も適したアーキテクチャを選定します。ただし、本節はこの比較を行わずに、Mask R-CNN を最適なアーキテクチャとして扱い、次のステップに進みます。
次のステップでは、訓練サブセットと検証サブセットを統合し、最適と判断したアーキテクチャを最初から訓練します。その準備として、まず訓練サブセットと検証サブセットを結合します。
!mkdir -p teethsegm/trainvalid/images
!mkdir -p teethsegm/trainvalid/masks
!cp teethsegm/train/images/* teethsegm/trainvalid/images
!cp teethsegm/valid/images/* teethsegm/trainvalid/images
!cp teethsegm/train/masks/* teethsegm/trainvalid/masks
!cp teethsegm/valid/masks/* teethsegm/trainvalid/masks
次に、モデルの構築を行います。先ほど可視化した検証性能の推移グラフを確認した結果、数エポックの訓練で十分に高い予測性能を達成できることがわかりました。そこで、ここでは訓練サブセットと検証サブセットを統合したデータを用いて、5 エポックのみ訓練を行います。
# model
model = maskrcnn(num_classes=1)
model.to(device)
# training parameters
optimizer = torch.optim.SGD(model.parameters(), lr=1e-3, weight_decay=1e-4)
lr_scheduler = torch.optim.lr_scheduler.StepLR(optimizer, step_size=3, gamma=0.1)
# training data
train_loader = torch.utils.data.DataLoader(
TeethDataset('teethsegm/trainvalid'),
batch_size=4, shuffle=True, collate_fn=lambda x: tuple(zip(*x)))
# training
num_epochs = 5
metric_dict = []
for epoch in range(num_epochs):
model.train()
epoch_loss_dict = {}
for images, targets in train_loader:
images = [img.to(device) for img in images]
targets = [{k: v.to(device) for k, v in t.items()} for t in targets]
batch_loss_dict = model(images, targets)
batch_tol_loss = 0
for loss_type, loss_val in batch_loss_dict.items():
batch_tol_loss += loss_val
if loss_type in epoch_loss_dict:
epoch_loss_dict[f'train_{loss_type}'] += loss_val.item()
else:
epoch_loss_dict[f'train_{loss_type}'] = loss_val.item()
optimizer.zero_grad()
batch_tol_loss.backward()
optimizer.step()
lr_scheduler.step()
# record training loss
epoch_loss_dict['train_total_loss'] = sum(epoch_loss_dict.values())
metric_dict.append({k: v / len(train_loader) for k, v in epoch_loss_dict.items()})
metric_dict[-1]['epoch'] = epoch + 1
print(metric_dict[-1])
Show code cell output
{'train_loss_classifier': 0.029262897041108873, 'train_loss_box_reg': 0.04783724413977729, 'train_loss_mask': 0.032386458582348294, 'train_loss_objectness': 0.0024690950910250344, 'train_loss_rpn_box_reg': 0.0052216748396555586, 'train_total_loss': 0.11717736969391505, 'epoch': 1}
{'train_loss_classifier': 0.024184823036193848, 'train_loss_box_reg': 0.04432510998513964, 'train_loss_mask': 0.025044288900163438, 'train_loss_objectness': 0.0019431029342942769, 'train_loss_rpn_box_reg': 0.0027278123630417716, 'train_total_loss': 0.09822513721883297, 'epoch': 2}
{'train_loss_classifier': 0.021655243304040697, 'train_loss_box_reg': 0.04186083210839166, 'train_loss_mask': 0.02072103652689192, 'train_loss_objectness': 0.0025216293417745167, 'train_loss_rpn_box_reg': 0.003110025698939959, 'train_total_loss': 0.08986876698003875, 'epoch': 3}
{'train_loss_classifier': 0.021162519852320354, 'train_loss_box_reg': 0.04163465234968397, 'train_loss_mask': 0.021362900733947754, 'train_loss_objectness': 0.002997058547205395, 'train_loss_rpn_box_reg': 0.0032064848475986058, 'train_total_loss': 0.09036361633075608, 'epoch': 4}
{'train_loss_classifier': 0.02135988904370202, 'train_loss_box_reg': 0.04249116447236803, 'train_loss_mask': 0.02216295235686832, 'train_loss_objectness': 0.0023081333686908088, 'train_loss_rpn_box_reg': 0.004040585623847114, 'train_total_loss': 0.0923627248654763, 'epoch': 5}
訓練が完了したら、訓練済みモデルの重みをファイルに保存します。
model.to('cpu')
torch.save(model.state_dict(), 'teethsegm.pth')
5.2.4. モデル評価#
最適なモデルが得られたら、次にテストデータを用いてモデルを詳細に評価します。
test_loader = torch.utils.data.DataLoader(
TeethDataset('teethsegm/test'),
batch_size=4, shuffle=True, collate_fn=lambda x: tuple(zip(*x)))
model = maskrcnn(num_classes=1, weights='teethsegm.pth')
model.to(device)
model.eval()
metric = torchmetrics.detection.mean_ap.MeanAveragePrecision()
iou = 0
n_targets = 0
with torch.no_grad():
for images, targets in test_loader:
images = [img.to(device) for img in images]
targets = [{k: v.to(device) for k, v in t.items()} for t in targets]
pred_targets = model(images)
metric.update(pred_targets, targets)
for i in range(len(targets)):
pred_mask = pred_targets[i]['masks'].squeeze(1).any(dim=0)
true_mask = targets[i]['masks'].any(dim=0)
iou += torchmetrics.functional.jaccard_index(pred_mask.unsqueeze(0), true_mask.unsqueeze(0), num_classes=1, task='binary')
n_targets += 1
metrics = [{k: v.cpu().detach().numpy().tolist()} for k, v in metric.compute().items()]
metrics.append({'avg_iou': iou.item() / n_targets})
metrics
[{'map': 0.3129052519798279},
{'map_50': 0.7395800948143005},
{'map_75': 0.16131183505058289},
{'map_small': -1.0},
{'map_medium': 0.02635670080780983},
{'map_large': 0.3330473303794861},
{'mar_1': 0.017307693138718605},
{'mar_10': 0.19769230484962463},
{'mar_100': 0.504230797290802},
{'mar_small': -1.0},
{'mar_medium': 0.26153847575187683},
{'mar_large': 0.5170040726661682},
{'map_per_class': -1.0},
{'mar_100_per_class': -1.0},
{'classes': 1},
{'avg_iou': 0.38422379493713377}]
5.2.5. 推論#
推論時にも、訓練時と同じように torchvision モジュールからアーキテクチャを呼び出し、出力層のクラス数を設定します。その後、load_state_dict メソッドを使って、訓練済みの重みファイルをモデルにロードします。これらの操作はすべて maskrcnn 関数で定義されているので、その関数を利用します。
model = maskrcnn(num_classes=1, weights='teethsegm.pth')
model.to(device)
model.eval()
Show code cell output
MaskRCNN(
(transform): GeneralizedRCNNTransform(
Normalize(mean=[0.485, 0.456, 0.406], std=[0.229, 0.224, 0.225])
Resize(min_size=(800,), max_size=1333, mode='bilinear')
)
(backbone): BackboneWithFPN(
(body): IntermediateLayerGetter(
(conv1): Conv2d(3, 64, kernel_size=(7, 7), stride=(2, 2), padding=(3, 3), bias=False)
(bn1): FrozenBatchNorm2d(64, eps=0.0)
(relu): ReLU(inplace=True)
(maxpool): MaxPool2d(kernel_size=3, stride=2, padding=1, dilation=1, ceil_mode=False)
(layer1): Sequential(
(0): Bottleneck(
(conv1): Conv2d(64, 64, kernel_size=(1, 1), stride=(1, 1), bias=False)
(bn1): FrozenBatchNorm2d(64, eps=0.0)
(conv2): Conv2d(64, 64, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1), bias=False)
(bn2): FrozenBatchNorm2d(64, eps=0.0)
(conv3): Conv2d(64, 256, kernel_size=(1, 1), stride=(1, 1), bias=False)
(bn3): FrozenBatchNorm2d(256, eps=0.0)
(relu): ReLU(inplace=True)
(downsample): Sequential(
(0): Conv2d(64, 256, kernel_size=(1, 1), stride=(1, 1), bias=False)
(1): FrozenBatchNorm2d(256, eps=0.0)
)
)
(1): Bottleneck(
(conv1): Conv2d(256, 64, kernel_size=(1, 1), stride=(1, 1), bias=False)
(bn1): FrozenBatchNorm2d(64, eps=0.0)
(conv2): Conv2d(64, 64, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1), bias=False)
(bn2): FrozenBatchNorm2d(64, eps=0.0)
(conv3): Conv2d(64, 256, kernel_size=(1, 1), stride=(1, 1), bias=False)
(bn3): FrozenBatchNorm2d(256, eps=0.0)
(relu): ReLU(inplace=True)
)
(2): Bottleneck(
(conv1): Conv2d(256, 64, kernel_size=(1, 1), stride=(1, 1), bias=False)
(bn1): FrozenBatchNorm2d(64, eps=0.0)
(conv2): Conv2d(64, 64, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1), bias=False)
(bn2): FrozenBatchNorm2d(64, eps=0.0)
(conv3): Conv2d(64, 256, kernel_size=(1, 1), stride=(1, 1), bias=False)
(bn3): FrozenBatchNorm2d(256, eps=0.0)
(relu): ReLU(inplace=True)
)
)
(layer2): Sequential(
(0): Bottleneck(
(conv1): Conv2d(256, 128, kernel_size=(1, 1), stride=(1, 1), bias=False)
(bn1): FrozenBatchNorm2d(128, eps=0.0)
(conv2): Conv2d(128, 128, kernel_size=(3, 3), stride=(2, 2), padding=(1, 1), bias=False)
(bn2): FrozenBatchNorm2d(128, eps=0.0)
(conv3): Conv2d(128, 512, kernel_size=(1, 1), stride=(1, 1), bias=False)
(bn3): FrozenBatchNorm2d(512, eps=0.0)
(relu): ReLU(inplace=True)
(downsample): Sequential(
(0): Conv2d(256, 512, kernel_size=(1, 1), stride=(2, 2), bias=False)
(1): FrozenBatchNorm2d(512, eps=0.0)
)
)
(1): Bottleneck(
(conv1): Conv2d(512, 128, kernel_size=(1, 1), stride=(1, 1), bias=False)
(bn1): FrozenBatchNorm2d(128, eps=0.0)
(conv2): Conv2d(128, 128, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1), bias=False)
(bn2): FrozenBatchNorm2d(128, eps=0.0)
(conv3): Conv2d(128, 512, kernel_size=(1, 1), stride=(1, 1), bias=False)
(bn3): FrozenBatchNorm2d(512, eps=0.0)
(relu): ReLU(inplace=True)
)
(2): Bottleneck(
(conv1): Conv2d(512, 128, kernel_size=(1, 1), stride=(1, 1), bias=False)
(bn1): FrozenBatchNorm2d(128, eps=0.0)
(conv2): Conv2d(128, 128, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1), bias=False)
(bn2): FrozenBatchNorm2d(128, eps=0.0)
(conv3): Conv2d(128, 512, kernel_size=(1, 1), stride=(1, 1), bias=False)
(bn3): FrozenBatchNorm2d(512, eps=0.0)
(relu): ReLU(inplace=True)
)
(3): Bottleneck(
(conv1): Conv2d(512, 128, kernel_size=(1, 1), stride=(1, 1), bias=False)
(bn1): FrozenBatchNorm2d(128, eps=0.0)
(conv2): Conv2d(128, 128, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1), bias=False)
(bn2): FrozenBatchNorm2d(128, eps=0.0)
(conv3): Conv2d(128, 512, kernel_size=(1, 1), stride=(1, 1), bias=False)
(bn3): FrozenBatchNorm2d(512, eps=0.0)
(relu): ReLU(inplace=True)
)
)
(layer3): Sequential(
(0): Bottleneck(
(conv1): Conv2d(512, 256, kernel_size=(1, 1), stride=(1, 1), bias=False)
(bn1): FrozenBatchNorm2d(256, eps=0.0)
(conv2): Conv2d(256, 256, kernel_size=(3, 3), stride=(2, 2), padding=(1, 1), bias=False)
(bn2): FrozenBatchNorm2d(256, eps=0.0)
(conv3): Conv2d(256, 1024, kernel_size=(1, 1), stride=(1, 1), bias=False)
(bn3): FrozenBatchNorm2d(1024, eps=0.0)
(relu): ReLU(inplace=True)
(downsample): Sequential(
(0): Conv2d(512, 1024, kernel_size=(1, 1), stride=(2, 2), bias=False)
(1): FrozenBatchNorm2d(1024, eps=0.0)
)
)
(1): Bottleneck(
(conv1): Conv2d(1024, 256, kernel_size=(1, 1), stride=(1, 1), bias=False)
(bn1): FrozenBatchNorm2d(256, eps=0.0)
(conv2): Conv2d(256, 256, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1), bias=False)
(bn2): FrozenBatchNorm2d(256, eps=0.0)
(conv3): Conv2d(256, 1024, kernel_size=(1, 1), stride=(1, 1), bias=False)
(bn3): FrozenBatchNorm2d(1024, eps=0.0)
(relu): ReLU(inplace=True)
)
(2): Bottleneck(
(conv1): Conv2d(1024, 256, kernel_size=(1, 1), stride=(1, 1), bias=False)
(bn1): FrozenBatchNorm2d(256, eps=0.0)
(conv2): Conv2d(256, 256, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1), bias=False)
(bn2): FrozenBatchNorm2d(256, eps=0.0)
(conv3): Conv2d(256, 1024, kernel_size=(1, 1), stride=(1, 1), bias=False)
(bn3): FrozenBatchNorm2d(1024, eps=0.0)
(relu): ReLU(inplace=True)
)
(3): Bottleneck(
(conv1): Conv2d(1024, 256, kernel_size=(1, 1), stride=(1, 1), bias=False)
(bn1): FrozenBatchNorm2d(256, eps=0.0)
(conv2): Conv2d(256, 256, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1), bias=False)
(bn2): FrozenBatchNorm2d(256, eps=0.0)
(conv3): Conv2d(256, 1024, kernel_size=(1, 1), stride=(1, 1), bias=False)
(bn3): FrozenBatchNorm2d(1024, eps=0.0)
(relu): ReLU(inplace=True)
)
(4): Bottleneck(
(conv1): Conv2d(1024, 256, kernel_size=(1, 1), stride=(1, 1), bias=False)
(bn1): FrozenBatchNorm2d(256, eps=0.0)
(conv2): Conv2d(256, 256, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1), bias=False)
(bn2): FrozenBatchNorm2d(256, eps=0.0)
(conv3): Conv2d(256, 1024, kernel_size=(1, 1), stride=(1, 1), bias=False)
(bn3): FrozenBatchNorm2d(1024, eps=0.0)
(relu): ReLU(inplace=True)
)
(5): Bottleneck(
(conv1): Conv2d(1024, 256, kernel_size=(1, 1), stride=(1, 1), bias=False)
(bn1): FrozenBatchNorm2d(256, eps=0.0)
(conv2): Conv2d(256, 256, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1), bias=False)
(bn2): FrozenBatchNorm2d(256, eps=0.0)
(conv3): Conv2d(256, 1024, kernel_size=(1, 1), stride=(1, 1), bias=False)
(bn3): FrozenBatchNorm2d(1024, eps=0.0)
(relu): ReLU(inplace=True)
)
)
(layer4): Sequential(
(0): Bottleneck(
(conv1): Conv2d(1024, 512, kernel_size=(1, 1), stride=(1, 1), bias=False)
(bn1): FrozenBatchNorm2d(512, eps=0.0)
(conv2): Conv2d(512, 512, kernel_size=(3, 3), stride=(2, 2), padding=(1, 1), bias=False)
(bn2): FrozenBatchNorm2d(512, eps=0.0)
(conv3): Conv2d(512, 2048, kernel_size=(1, 1), stride=(1, 1), bias=False)
(bn3): FrozenBatchNorm2d(2048, eps=0.0)
(relu): ReLU(inplace=True)
(downsample): Sequential(
(0): Conv2d(1024, 2048, kernel_size=(1, 1), stride=(2, 2), bias=False)
(1): FrozenBatchNorm2d(2048, eps=0.0)
)
)
(1): Bottleneck(
(conv1): Conv2d(2048, 512, kernel_size=(1, 1), stride=(1, 1), bias=False)
(bn1): FrozenBatchNorm2d(512, eps=0.0)
(conv2): Conv2d(512, 512, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1), bias=False)
(bn2): FrozenBatchNorm2d(512, eps=0.0)
(conv3): Conv2d(512, 2048, kernel_size=(1, 1), stride=(1, 1), bias=False)
(bn3): FrozenBatchNorm2d(2048, eps=0.0)
(relu): ReLU(inplace=True)
)
(2): Bottleneck(
(conv1): Conv2d(2048, 512, kernel_size=(1, 1), stride=(1, 1), bias=False)
(bn1): FrozenBatchNorm2d(512, eps=0.0)
(conv2): Conv2d(512, 512, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1), bias=False)
(bn2): FrozenBatchNorm2d(512, eps=0.0)
(conv3): Conv2d(512, 2048, kernel_size=(1, 1), stride=(1, 1), bias=False)
(bn3): FrozenBatchNorm2d(2048, eps=0.0)
(relu): ReLU(inplace=True)
)
)
)
(fpn): FeaturePyramidNetwork(
(inner_blocks): ModuleList(
(0): Conv2dNormActivation(
(0): Conv2d(256, 256, kernel_size=(1, 1), stride=(1, 1))
)
(1): Conv2dNormActivation(
(0): Conv2d(512, 256, kernel_size=(1, 1), stride=(1, 1))
)
(2): Conv2dNormActivation(
(0): Conv2d(1024, 256, kernel_size=(1, 1), stride=(1, 1))
)
(3): Conv2dNormActivation(
(0): Conv2d(2048, 256, kernel_size=(1, 1), stride=(1, 1))
)
)
(layer_blocks): ModuleList(
(0-3): 4 x Conv2dNormActivation(
(0): Conv2d(256, 256, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))
)
)
(extra_blocks): LastLevelMaxPool()
)
)
(rpn): RegionProposalNetwork(
(anchor_generator): AnchorGenerator()
(head): RPNHead(
(conv): Sequential(
(0): Conv2dNormActivation(
(0): Conv2d(256, 256, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))
(1): ReLU(inplace=True)
)
)
(cls_logits): Conv2d(256, 3, kernel_size=(1, 1), stride=(1, 1))
(bbox_pred): Conv2d(256, 12, kernel_size=(1, 1), stride=(1, 1))
)
)
(roi_heads): RoIHeads(
(box_roi_pool): MultiScaleRoIAlign(featmap_names=['0', '1', '2', '3'], output_size=(7, 7), sampling_ratio=2)
(box_head): TwoMLPHead(
(fc6): Linear(in_features=12544, out_features=1024, bias=True)
(fc7): Linear(in_features=1024, out_features=1024, bias=True)
)
(box_predictor): FastRCNNPredictor(
(cls_score): Linear(in_features=1024, out_features=2, bias=True)
(bbox_pred): Linear(in_features=1024, out_features=8, bias=True)
)
(mask_roi_pool): MultiScaleRoIAlign(featmap_names=['0', '1', '2', '3'], output_size=(14, 14), sampling_ratio=2)
(mask_head): MaskRCNNHeads(
(0): Conv2dNormActivation(
(0): Conv2d(256, 256, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))
(1): ReLU(inplace=True)
)
(1): Conv2dNormActivation(
(0): Conv2d(256, 256, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))
(1): ReLU(inplace=True)
)
(2): Conv2dNormActivation(
(0): Conv2d(256, 256, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))
(1): ReLU(inplace=True)
)
(3): Conv2dNormActivation(
(0): Conv2d(256, 256, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))
(1): ReLU(inplace=True)
)
)
(mask_predictor): MaskRCNNPredictor(
(conv5_mask): ConvTranspose2d(256, 256, kernel_size=(2, 2), stride=(2, 2))
(relu): ReLU(inplace=True)
(mask_fcn_logits): Conv2d(256, 2, kernel_size=(1, 1), stride=(1, 1))
)
)
)
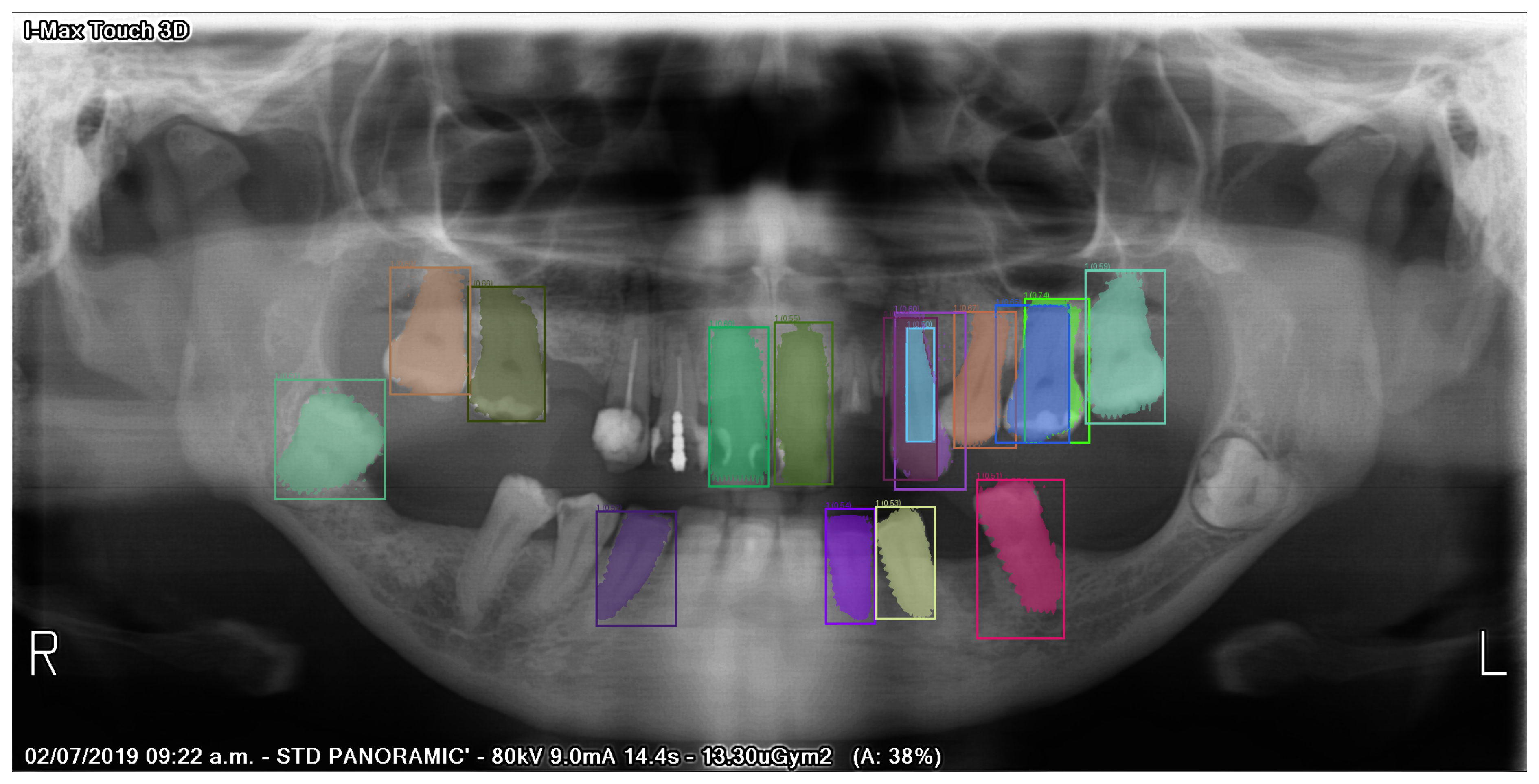
このモデルを利用して推論を行います。まず、1 枚の画像を指定し、PIL モジュールを用いて画像を開き、テンソル形式に変換した後、モデルに入力します。モデルは予測結果としてバウンディングボックスの座標(bboxes)、マスク(masks)、分類ラベル(labels)、および信頼スコア(scores)を出力します。ただし、信頼スコアが 0.5 未満のバウンディングボックスは採用せず、信頼スコアが高い結果のみを選択して利用します。
threshold = 0.5
image_path = 'teethsegm/test/images/13.jpg'
image = PIL.Image.open(image_path).convert('RGB')
input_tensor = torchvision.transforms.v2.functional.to_tensor(image).unsqueeze(0).to(device)
with torch.no_grad():
predictions = model(input_tensor)[0]
bboxes = predictions['boxes'][predictions['scores'] > threshold]
masks = predictions['masks'][predictions['scores'] > threshold]
labels = predictions['labels'][predictions['scores'] > threshold]
scores = predictions['scores'][predictions['scores'] > threshold]
検出されたオブジェクトのバウンディングボックスを入力画像に描画します。その後、PIL および matplotlib ライブラリを使用して、画像とその検出結果を可視化します。
image = PIL.Image.open(image_path).convert('RGBA')
overlay = PIL.Image.new('RGBA', image.size, (255, 255, 255, 0))
overlay_draw = PIL.ImageDraw.Draw(overlay)
for bbox, mask, label, score in zip(bboxes, masks, labels, scores):
col = tuple([random.randint(0, 255) for _ in range(3)]) + (128,)
# bbox
x1, y1, x2, y2 = bbox
draw = PIL.ImageDraw.Draw(image)
draw.rectangle(((x1, y1), (x2, y2)), outline=col[:3], width=3) # BBox is not transparent
draw.text((x1, y1 - 10), f'{label.item()} ({score:.2f})', fill=col[:3])
# mask
mask = mask.squeeze(0).cpu().numpy()
contours = skimage.measure.find_contours(mask, 0.5)
for contour in contours:
contour = np.flip(contour, axis=1).astype(int)
polygon = [tuple(point) for point in contour]
overlay_draw.polygon(polygon, fill=col)
blended_image = PIL.Image.alpha_composite(image, overlay)
fig = plt.figure()
ax = fig.add_subplot()
ax.imshow(blended_image)
ax.axis('off')
plt.show()